人工智能在传染病和抗菌药物耐药性方面的挑战与应用
作者:de la Fuente-Nunez, Cesar
近年来,人工智能(AI)极大地加速了新抗生素的发现
1,2,3,4,5。使用传统方法需要数年时间才能发现新的抗生素,而使用计算机现在可以在数小时内完成。尽管科学技术取得了这些进步,但每年仍有数百万人死于感染。
抗生素的过度使用和滥用导致泛耐药微生物的出现,仅2019年就导致超过400万人死亡6,7。然而,抗菌治疗未能有效治愈患者并根除病原体并不仅仅归因于抗生素耐药基因。多种因素,包括脓毒症等关键临床病症背后的复杂机制,导致抗生素失效(图 1)。1)。这个多方面的问题已成为全球日益关注的问题6,8,9。
最近,医学和生物数据的大幅增长刺激了许多基于人工智能的技术的发展。这些技术使计算机能够学习和适应日益复杂的信息10,11,12,13,14。在抗生素失效的背景下,人工智能为疾病诊断和药物发现提供了巨大的机会,特别是在传统抗生素无效的情况下15,16,17 号,18,19,20,21,22 号。
机器学习 (ML) 是人工智能的一个分支,在计算生物学、语言处理、游戏和计算机视觉等领域的多种应用中取得了重大进展。一些机器学习模型利用深度学习 (DL),这是一种利用深度神经网络 (DNN) 提取隐藏特征并预测复杂数据集(包括图像和生物分子结构)结果的方法5,10,23,24,25,26。
尖端算法已证明能够分析广泛的临床和实验数据,为疾病诊断、治疗预后以及预测或生成新型有效的抗菌化合物提供见解。这些计算机辅助技术有可能解决由抗生素无效引起的临床病症,而及时、准确的干预措施至关重要。
在这篇综述中,我们探讨了计算机引导方法在诊断、治疗方法和药物发现中的使用,并提供了各个层面的示例。我们重点介绍这些方法的技术特点和优势,展示它们如何提高患者护理和治疗开发的效率。此外,我们指出了与这些技术相关的局限性和挑战,包括数据可用性和道德问题,强调需要标准化数据集和当局干预以确保数据保护。
抗生素失效:一个被低估的全球健康问题
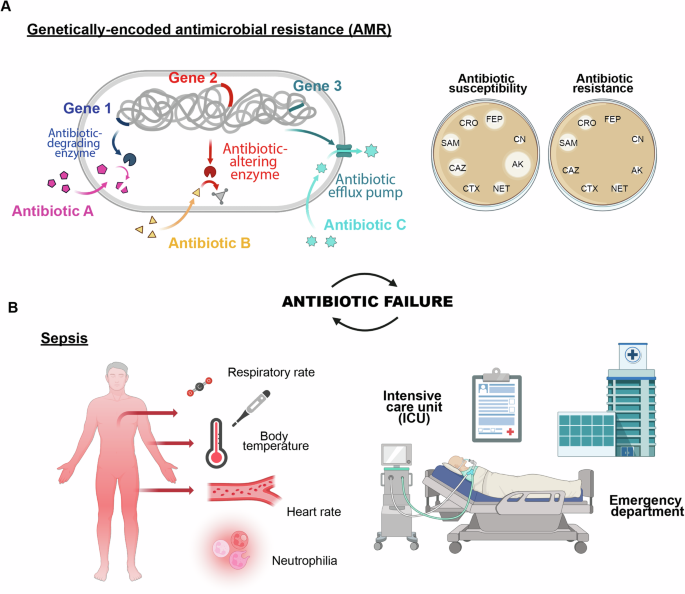
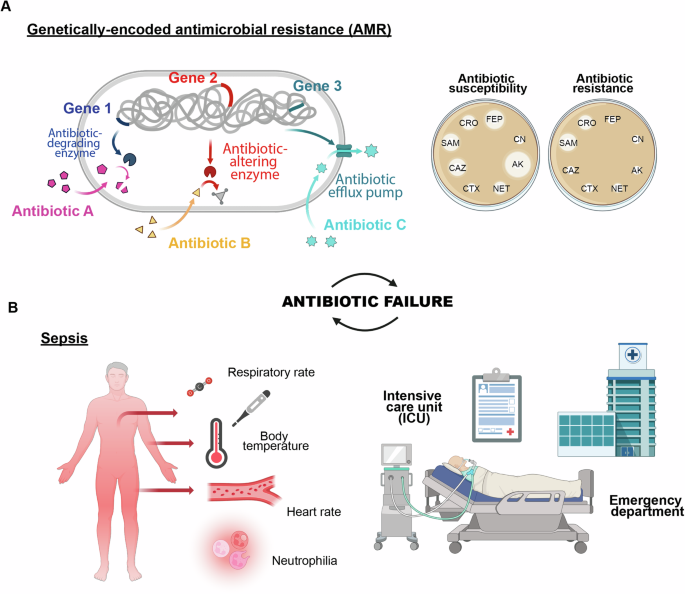
抗生素失败的主要原因之一是抗生素耐药表型的自然选择。2022 年 1 月发布的最新报告显示,六种主要细菌病原体,包括大肠杆菌,金黄色葡萄球菌,肺炎克雷伯菌,肺炎链球菌,鲍曼不动杆菌, 和铜绿假单胞菌,导致全球数百万人因抗菌素耐药性 (AMR) 死亡或与之相关7。美国疾病控制与预防中心 (CDC) 强调住院期间发生的耐药性感染显着且令人震惊地增加,将抗菌素耐药性视为当今人类健康的主要威胁之一27 号。2021 年,有 150 万人直接死于抗菌素耐药性,471 万人因此死亡6。根据最近提出的一个场景柳叶刀预计到 2050 年,AMR 造成的死亡人数将达到 191 万,相关死亡人数将达到 822 万。这表明,如果不采取进一步干预措施,到 2030 年 AMR 死亡率将不可能实现 10% 的降低(图 1)。1A)6。
除了耐药性进化的挑战之外,新药推向市场的过程平均需要 15 年。该持续时间涵盖药物发现和开发、临床前和临床试验、美国食品和药物管理局 (FDA) 的监管审查以及上市后安全监测等阶段28。其他担忧包括将新化合物推向市场所需的估计成本为 1.61 亿美元至 45 亿美元29。事实上,传统的药物发现方法通常涉及昂贵且耗时的高通量筛选,其评估各种细菌菌株的多种潜在化合物组合。因此,抗生素耐药菌株的持续出现,加上针对耐药物种的新的有效抗生素的缺乏,给医疗保健系统带来了相当大的压力。这引发了人们对整合最先进的人工智能模型和其他计算方法的兴趣,以加快识别具有所需特性的新药,同时探索更广泛的化学空间。
基因决定的抗菌药物耐药性,导致选择耐甲氧西林等菌株金黄色葡萄球菌(MRSA),万古霉素耐药肠球菌属(VRE),耐碳青霉烯类不动杆菌属和产生超广谱 β-内酰胺酶 (ESBL)肠杆菌目,只是导致抗生素失败的因素之一。抗生素失效被定义为抗菌治疗不再有效根除细菌感染,导致临床状况持续存在或恶化的任何事件9。虽然 AMR 病例以及抑菌活性和药物灭活的机制与抗生素的性能密切相关,但病原体定植(例如生物膜形成)和身体反应(例如脓毒症)等其他因素也受到抗生素的影响。感染类型和患者的临床状况。这些因素同样会导致抗生素失效(图 1)。1B)8,9。
细菌生物膜是包裹在聚合物基质中的微生物菌落,常见于表面,与约 65% 的感染有关,包括与导管和植入物等医疗设备相关的感染。由于抗生素扩散受限和基因表达改变等因素,生物膜细胞对抗生素的耐药性比浮游细胞高 10 至 1,000 倍30。此外,生物膜可以逃避宿主防御,使其持续存在并导致慢性感染30。在这种情况下,及时检测能够形成生物膜的细菌种类对于有效的治疗和管理至关重要8。
脓毒症是一种危急病症,是指对感染的免疫反应失控,最严重的情况涉及多个器官功能障碍。仅在美国,每年就有约 170 万人患败血症,其中约 35 万人在住院期间死亡31。尽管这些数字与 AMR 造成的预计死亡人数无法比较,但死于脓毒症的人数占全球每年报告的死亡人数的 19.7%9。例如,抗生素是治疗脓毒症的主要方法,但其有效性受到高死亡率(23-35%)和临床并发症的限制。长期或不当使用抗生素可能导致耐药性感染和不良副作用32。脓毒症的初始症状通常是非特异性的,从而延迟了致病病原体的识别和适当治疗的开始9。因此,初始治疗往往是经验性的、广谱的,导致过度接触非特异性抗菌治疗,这就是为什么它们在超过 20% 的病例中无效的原因(图 1)。1B)32。
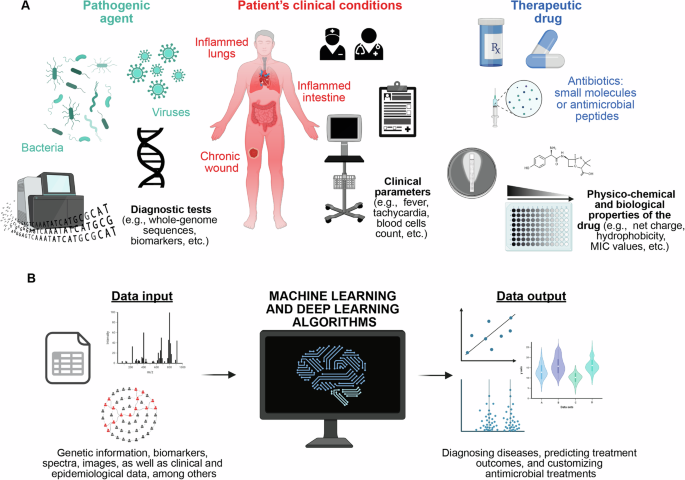
在这种背景下,基于人工智能的方法已成为分析广泛数据集并揭示人类观察不到的复杂模式的宝贵工具(图 1)。2A)。这有助于先进诊断工具和个性化治疗计划的开发(图1)。2B)33。
尽管人工智能在医疗诊断方面仍处于初级阶段,但不断增加的数据可用性为其更广泛的应用铺平了道路,特别是在检测癌症等疾病方面
33。在传染病和脓毒症的具体情况下,潜在机制和症状的复杂性对开发早期诊断工具提出了挑战。然而,机器学习模型通过工作流程管理、任务自动化和决策辅助在诊断方面有望发挥作用。此外,采用卷积神经网络 (CNN)、循环神经网络 (RNN) 和数据挖掘的深度学习框架在识别广泛数据集中的疾病模式方面日益发挥着重要作用34。这种能力在医疗保健系统内诊断、预测和分类疾病方面具有巨大的潜力。表总结了所选人工智能模型及其在诊断中的潜在应用的比较1。
在重症监护病房 (ICU) 的危重环境中,由于患者的紧迫性,图像、数字和文本等多种数据格式需要快速、准确的评估。此外,破译复杂的非线性数据关系至关重要。虽然线性回归等传统统计工具通过推荐“最佳拟合线”来通过数学方程提供见解,但深度学习方法能够细致地分析证据,而无需简化复杂的关系。深度学习可以同时处理多个输入,从而能够创建针对特定结果的预测模型。
例如,Steenkiste 等人。应用双向长短期记忆(LSTM)模型来预测 ICU 患者的血培养结果35。LSTM 是 RNN 的一种,这意味着模型的输入既是当前特征也是之前的状态,因此非常适合时间序列数据。具体来说,LSTM 包含一个模块来决定从过去的状态中记住和忘记什么,从而提高它们跟踪长期依赖性的能力。他们的模型使用了九个临床特征,包括体温、血小板计数、白细胞计数、C反应蛋白浓度、脓毒症相关器官衰竭评估、心率、呼吸频率、凝血酶原时间国际标准化比值和平均全身动脉压,进行了评估随着时间的推移,使用包含 2,177 名 ICU 患者的高质量数据库。该网络的接受者操作特征 (ROC) 曲线下面积(表示诊断性能)为 0.99,精确回忆曲线下平均面积 (AUC) 为 0.82,反映了其准确性。需要注意的是,ROC 曲线提供了一种可视化表示,通过绘制不同阈值的真阳性率(灵敏度)与假阳性率(1-特异性)来显示分类模型在各种阈值设置上的执行情况;另一方面,AUC 平均模型在区分正面和负面案例方面的表现,提供模型整体有效性的全面视图。除了显着的 ROC 和 AUC 值之外,Steenkiste 等人的研究结果还表明,在事件发生前几个小时进行预测只会导致预测能力略有下降35。
人工智能在微生物学中的进一步应用可能涉及处理图像、质谱和细菌全基因组序列等数据16,36。
史密斯等人。采用预先训练的 CNN,最初设计用于图像分类,并对其进行调整以识别各种细菌,包括不同的革兰氏染色形态(例如,革兰氏阳性细菌)球菌呈簇状排列,革兰氏阳性球菌呈链状排列的细菌和革兰氏阴性杆菌)从血培养样本中检测呈阳性。CNN 使用应用于图像和各种尺度的学习滤波器组来提供用于分类或其他任务的丰富特征。总共大约 100,000 个分类图像部分(革兰氏染色载玻片的裁剪部分)被输入到 CNN37。该网络在对图像作物进行分类时达到了约 95% 的准确率,在对所有类别的整个幻灯片进行分类时达到了 92.5% 的准确率37。
另一个例子是 Zagajewski 等人最近的工作,他们使用 CNN 在单细胞水平上区分易感表型和耐药(未经治疗)表型,从而促进对抗生素表现出不同生理反应的单个细胞的快速和详细的表型分析38。
相反,大多数利用质谱的人工智能应用侧重于将未知样品的蛋白质组指纹(表示为质荷 (m/z) 峰值和强度)与已知光谱的精选数据库进行比较。传统软件使用预定义的规则来评估未知谱与数据库条目之间的相似性。相比之下,机器学习方法利用内在数据模式来开发预测模型。这种方法对于预测抗菌素耐药性和进行菌株分型或爆发调查特别有效,无需显式编程16,39,40。例如,韦斯等人。收集临床菌株的质谱数据并将其与耐药性概况配对,建立广泛的 DIAMS 数据集10,41。该数据集包括 70 多种抗菌药物的耐药性信息以及 803 种病原体的 300,000 多个临床菌株的质谱数据。使用该数据集训练了三种机器学习算法:逻辑回归、深度神经网络分类器(多层感知器,MLP)和梯度增强决策树(LightGBM)。其中,MLP 和 LightGBM 成为最有效的分类器。这种创新方法展示了以高效可靠的方式显着改善抗生素管理和优化感染治疗策略的潜力41。
AI 和 ML 方法还利用细菌 DNA 序列及其抗菌敏感性表型42。事实上,机器学习分析了这些细菌分离株的大型库,研究了它们的 DNA 序列和抗菌素耐药性之间的联系。采用各种 ML 算法,例如分析 DNA 短序列 (k-mers) 并根据其对抗菌素耐药性表型的预测值对其进行加权的算法42。一旦开发出这些模型,他们就可以在测序过程中可靠地预测新分离株的敏感性。
此外,关于 ML 模型的令人鼓舞的发现不仅能够预测表型特征,还能预测所评估的抗菌药物观察到的最低抑菌浓度 (MIC)。例如,Nguyen 等人。使用基于极端梯度增强 (XGBoost) 的 ML 模型,成功预测了非伤寒患者的耐药性和 MIC沙门氏菌43。此外,结合基因组和转录组还可以增强基于机器学习的分类器在预测抗菌素耐药性表型方面的预测能力,特别是在革兰氏阴性 ESKAPE 病原体中42。
除了高精度之外,模型的可解释性对于获得患者和临床医生对人工智能的信任和接受也至关重要。可解释性方法分为两大类:本质上可解释的模型(或“白盒”模型)和“黑盒”模型的事后解释。在临床环境中,通常首选可直接解释的模型,因为它们很容易被所有利益相关者理解44,45。然而,对于更复杂的数据,可能需要事后方法。例如,Drouin 等人。开发了一种稀疏的、基于规则的模型,称为集合覆盖机 (SCM),利用基因组 k-mers 来预测抗菌药物耐药性46。另一方面,马丁内斯-阿古罗等人。将各种循环神经网络(RNN)与事后 Shapley 附加解释(SHAP)相结合,利用 ICU 电子健康记录来早期预测抗菌药物耐药性47。SHAP 是一种与模型无关的局部解释工具,根据特征与个体预测的相关性对特征进行排名,并且可以应用于多种类型的模型48。尽管它很实用,但关于事后人工智能可解释性方法仍然存在许多悬而未决的问题,因为它们本质上是底层模型的不完美表示。必须根据具体情况仔细考虑其使用的权衡49。
未来,预计人工智能工具的数量、人工智能软件分析的精确度和可靠性、人工智能与临床微生物学实验室协议的集成以及模型可解释性的进步都将继续扩大。
人工智能用于传染病治疗
在临床环境中,迫切需要一种快速且易于使用的方法来预测细菌抗菌药物敏感性,以制定明智的处方,并且人工智能可以通过分析数据(例如遗传学、生活方式和生物标志物)、预测结果来帮助推进个性化治疗。有针对性的干预和优化临床策略50。
例如,坎吉拉尔等人。利用 2007 年至 2013 年在波士顿记录的 10,053 名单纯性尿路感染 (UTI) 患者的电子健康记录开发了一个 ML 模型,提出了最精确的抗生素51。对 2014 年至 2016 年间在同一家医院就诊的 3629 名无并发症尿路感染患者进行回顾性测试,该算法的表现优于临床医生,将二线抗生素的使用量减少了 67%51。
Yelin 等人展示了另一个例子。他开发了一种机器学习算法,能够根据患者人口统计数据、尿培养历史和药品购买记录个性化抗生素处方52。他们使用跨越十年的纵向数据,检查了一个包含 711,099 个非医院获得性尿路感染实例的数据集,涉及 315,047 名个体患者。开发的算法可有效预测个性化的针对特定药物的抗菌药物耐药性,从而将治疗不匹配率从超过 8% 降低至 6% 以下52。
人工智能在医疗保健环境中的另一个应用是聊天机器人,它通过自然的、类人的语言交互将患者与信息联系起来。在传染病的背景下,聊天机器人可以在及时提供有关症状、预防和治疗方案的信息方面发挥至关重要的作用53。聊天机器人通常由四个核心模块组成:文本理解模块、对话管理器、文本生成组件以及用于培训和操作使用的数据库层13。文本理解模块使用模式匹配 [关键字/字符串匹配和可扩展标记语言 (XML) 变体,如人工智能标记语言 (AIML)]、机器学习(决策树和随机森林)和自然语言处理等方法来解释用户输入。NLP)技术(如命名实体识别)13。一些聊天机器人还使用基于规则的系统或固定输入方法,而混合方法则结合了多种技术。例如,像 Google 的 Dialogflow 这样的 Web 服务将机器学习与基于规则的方法结合起来使用54,55。对话管理模块处理用户输入并管理对话,将输入链接到适当的响应。采用两种主要的数据管理类型:静态管理(使用模式匹配或基于规则的系统)和动态管理(利用机器学习或 Web 服务进行上下文切换和意图识别)13。大多数聊天机器人采用三种类型的数据存储库中的一种或多种: 医学知识存储库(来自本地或来自维基百科等在线平台)18,56,用户数据存储用于响应定制57,58,以及对话脚本17 号,常用于模式匹配。通过将聊天机器人整合到传染病的管理中,医疗保健提供者可以加强患者教育、改善信息获取并简化沟通,最终带来更好的健康结果。在整个 COVID-19 大流行期间,疾病控制与预防中心 (CDC) 和世界卫生组织 (WHO) 等机构都采用聊天机器人来分发信息、促进安全实践并提供情感支持。例如,CDC 使用机器学习和自然语言处理开发了“Clara”14。Clara 被称为“冠状病毒自我检查器”,旨在帮助个人在遇到潜在的 COVID-19 症状时确定适当的行动。该聊天机器人是与 CDC 基金会和 Microsoft 的 Azure Healthcare Bot 服务合作创建的14,59。
道德、数据隐私和算法偏见
尽管人工智能在医疗保健领域的应用前景广阔,但它也带来了重大的伦理挑战,例如(1)获得数据使用的知情同意,(2)确保安全性和透明度,(3)保护数据隐私,(4)解决算法公平性和偏见60。事实上,随着人工智能成为个性化医疗不可或缺的一部分,保护患者数据和确保公平至关重要。大量敏感健康信息的收集需要严格的数据隐私措施,例如加密和受控访问,以防止未经授权的使用61。当局正在努力为人工智能的安全、道德部署制定明确的指导方针,医疗保健专业人员、开发人员和政策制定者之间的持续合作对于引导人工智能在医学中不断发展的作用至关重要。美国 FDA 和英国药品和保健产品监管局 (MHRA) 等监管机构负责监控人工智能在医疗保健中的使用,确保遵守安全、隐私和质量标准62。他们进行审计和检查,以确保利益相关者遵循必要的准则。《通用数据保护条例》(GDPR) 规定了严格的数据隐私义务,而自 2024 年 1 月起生效的《欧洲数据法》则规范数据(包括健康数据)的公平访问和使用。与此同时,世界卫生组织强调透明度、安全性和公平性等原则,指导各国制定法规,确保人工智能在医疗保健领域的道德部署,并促进监督和问责机制62。
此外,人工智能模型必须使用多样化的、有代表性的数据来开发,以避免医疗保健领域长期存在偏见。医疗保健领域的公平是指为所有患者提供公平的护理、资源和结果,无论其背景或社会地位如何。在人工智能驱动的医疗保健中,实现公平需要开发没有偏见的算法,并且能够为所有患者群体提供准确的诊断和治疗63。用于训练人工智能的数据、算法的设计以及医疗保健专业人员和患者与人工智能系统交互的方式都可能产生偏差。为了应对这些挑战,必须使用多样化且具有代表性的数据,进行定期审核,并向临床医生和患者提供有关潜在偏见以及如何应对这些偏见的教育63。
人工智能用于药物发现
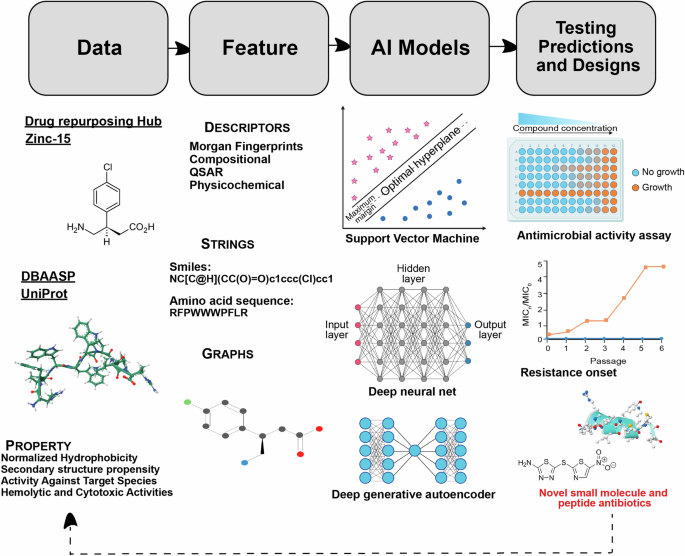
除了在诊断和传染病治疗中的应用之外,基于人工智能的方法还彻底改变了我们预测生物分子特性和结构的能力,同时还能够生成新的活性化合物(图 1)。3)11,21,22 号,64。此外,机器学习驱动的建模提供了绕过与传统药物发现方法相关的某些限制的途径,促进抗菌分子的快速设计和应用15,19,23,65,66,67,68。随着抗菌药物耐药性物种的日益扩散以及开发新药所面临的固有挑战,这两种情况都会极大地导致抗生素治疗的失败,因此迫切需要加快新型抗菌药物的设计2,10,19,20,21,22 号,69。
抗菌肽(AMP)和小分子均可作为治疗细菌感染的治疗剂。然而,传统高通量实验筛选的时间限制以及毒性、稳定性差、细胞渗透有限和合成成本等挑战阻碍了它们的鉴定和转化为临床应用。70,71。AMP 和小分子药物在临床审批上都存在障碍。然而,小分子合理设计背后的科学更加成熟,因此它们构成了绝大多数已批准的抗生素。天然 AMP 因其多样化的结构和功能、强大的抗菌特性以及极小的诱导耐药性的倾向,越来越多地被认为是传统抗生素的有前途的替代品和抗菌治疗的潜在继任者。因此,合成设计的 AMP 作为未来的治疗药物也具有广阔的前景72,73,基于 AMP 的抗生素(例如多粘菌素 B 和杆菌肽)也在临床上常用74,75。
机器学习方法已用于生物活性预测数十年23,76,77,78,79,80。这些技术被广泛地称为定量结构-活性关系(QSAR)模型。使用的底层机器学习模型可能会有所不同[例如,支持向量机(SVM)、朴素贝叶斯、神经网络等],但该框架涉及学习某些输入特征(通常是分子或肽描述符)和活性效力(或更一般地说,任何化学性质)(图 1)3)。为了对抗 AMR 和识别新的潜在药物,预测 ML 模型已被证明可以有效预测两种小分子抗生素的生物活性5,20,81和 AMP22 号,79,82。它们还可用于预测与候选药物相关的次要特性,例如溶解度、毒性等。83。
最近,已经开发出依赖自动学习特征而不是预定义分子描述符的深度学习模型来预测小分子和肽的各种特性。通常,这些编码字符串表示形式(例如 SMILES、SELFIES84将分子或肽的(氨基酸序列)放入降维的潜在嵌入空间中,然后可以将其用于训练预测器。图神经网络(GNN)也是分子特性预测模型的热门选择85。这些将分子的原子和键表示为图,并且可以从节点的隐藏状态(以及可选地从边缘)推断全局属性。GNN可以考虑对分子的三维特征进行建模。除字符串和图形外,DL模型还可以考虑其他形式的输入表示形式,包括预定义的描述符,图像,自然语言文本描述和频谱图(图。3)。这些模型的体系结构范围可以从卷积网,复发网(例如,长期短期内存网络86),传递神经网以及基于注意力的方法(例如变压器编码器)86。预测模型甚至已与图形解释方法结合使用,以识别负责某些特征的子结构,从而深入了解其他不透明的DL方法(图。3)21,87。
当要找到具有抗菌特性的药物时,这些预测模型可用于在硅虚拟筛选非常大的数据库中,因为其推断速度与湿式LAB筛选或基于物理学的评分相比。例如,这使得可以搜索大量的基因组和蛋白质组学数据以获取可能的AMP73,86。筛选数十亿个可能的6-9个氨基酸肽中的每一个甚至是可行的77。可以通过重组或化学过程在实验室中从自然或灭绝生物中采购放大器,也可以在实验室中合成。超过2,603种肽抗生素已在人类蛋白质组中进行了计算鉴定73,78,88,虽然在人类肠道宏基因组中发现了另外323种肽抗生素编码89。对全球微生物组进行了广泛的计算分析,其中包括63,410个宏基因组和87,920个微生物基因组,导致在微生物暗物质中发现了近100万个新的抗生素分子,其中一些在链锁小鼠模型中证明了疗效。这项最近的研究代表了迄今为止对抗生素发现的生物学数据的最大探索78。从假说,即这种抗生素肽不仅存在于人类蛋白质组中,而且存在于整个生命之树中,Maasch等人。应用机器学习模型来搜索我们最亲密的尼安德特人和丹尼索牛丝中的类似分子,从而有效地启动了分子去灭绝的领域22 号,90。在这项工作的基础上,团队开发了一种新的深度学习模型,称为Apex,以挖掘所有已知灭绝的生物用于抗生素22 号。这种方法导致在整个进化史中发现了抗生素,包括来自羊毛猛mm和其他生物体的临床前候选者。值得注意的是,在灭绝的生物中发现了超过37,000个序列。这些加密的肽中的几个以纳摩尔浓度协同起作用,以靶向病原体,并在临床前小鼠模型中表现出有效的抗感染活性22 号,90。
同样值得一提的是,在现有和潜在的药物分子库中鉴定小分子抗生素,Stokes等人的类似努力,并成功地报道了重新塑造药物分子的抗生素活性,其中一种证明了广谱活性。作者在2335个分子上训练了通知的神经网络(MPNN),以预测二进制大肠杆菌抑制作用(ROC 0.896),并将其应用于已知分子,用于重新定位或命名。MPNN是一种GNN的类型,它通过使用来自相邻节点的信息更新当地状态并汇总这些信息以提供全球表示形式。然后在体外测试具有最佳预测活性的分子,其中99个显示抑制作用中的51个。随后,刘等人。训练了类似的筛查模型鲍曼不动杆菌使用7684个实验测试分子的数据集(ROC 0.792),抗生素候选物4,5,91。他们还将该模型应用于一组重新利用的候选药物,并报告240个经验验证的分子中有40个显示出> 80%的生长抑制作用。总而言之,与传统的高通量筛查相比,该技术可以减少筛查时间,并且对于药物重新塑造特别有用。
为了发现新物质,过去几年中越来越多地使用了生成深度学习模型。生成模型依赖于对训练数据的分布进行建模,然后从该学到的分布中对新分子进行采样。这些可以使用标记的数据,未标记的数据或两者的组合来训练这些。如果该模型采用不需要(例如活动)标签的无监督或自我监督的学习,则可能会在类似药物的大分子数据库上培训模型(例如Zinc-1592)或生物活性肽(例如Uniprot93),因此在世代的多样性和新颖性方面表现出了强劲而改善的表现94。像预测模型一样,这些模型可以使用不同的输入表示,例如图形或字符串。流行的生成体系结构是自动回归解码器,编码器架构体系结构和生成对抗网络。最终分子的产生可以通过结合已知片段来建立结构95或直接以复发或自回旋方式生成输出(例如使用VAE69,96,gnns97,或变压器98,99,100)。
本身,无条件的生成模型只会再现训练数据分布,而训练数据分布不保证与一组用户选择的标准(活动,毒性等)定义的目标分布相匹配。Capecchi等人使用的一种简单方法。为了产生非溶血的放大器是通过对目标分布进行转移学习来微调广泛训练的模型。在那里,作者在整个DBAASP上训练了一个RNN,然后对两种型号进行微调,一种仅在非溶血性肽上进行活跃P.铜绿/a。鲍曼不动杆菌另一个是那些活跃的人金黄色葡萄球菌79。最终,对28肽进行了合成和测试,其中8种是活性和非溶血性的79。或者,来自(集合)预测模型的其他反馈或奖励信号通常用于指导生成模型的生成。存在许多具有特定特性的人工制品的有条件或受控生成的算法和方法。在这里,选择是广泛的。蒙特卡洛抽样20,69,101以及有条件的VAE和生成对抗网络(GAN)102是常见的方法。例如,Das等人。在生物活性和未标记的肽序列上以无监督的方式培训了正规的变异自动编码器,然后在学习的潜在空间上进行了属性条件采样,并从LSTM模型中进行了其他毒性过滤69。VAE将输入编码为压缩概率潜在空间,然后可以用来通过采样和解码来生成类似于训练的新数据。有条件的采样使该模型可以从较大的潜在空间内的特定子分布中汲取。这种方法在不利用任何现有的AMP原型作为生成种子的情况下,成功发现20个AI设计的肽中有7种表现出低至高抗菌活性。尤其重要的是,经过验证的AI设计的AMP中有两个确实显示出广泛的功效,体外和体内毒性较低。此外,这两个AI发现的肽对针对肺炎克雷伯菌对多粘蛋白B的耐药性,这是一种抗生素的抗生素。他们还减轻了耐药性的发作大肠杆菌。在该研究中,基于生成DL的方法在发现具有所需特性的新型AMP时产生了10%的成功率和48天的快速周转。同样,Swanson等人最近采用了使用Monte-Carlo树搜索来设计活跃的分子的生成AI鲍曼不动杆菌20。他们确定了六种抑制生长的新化合物鲍曼不动杆菌以及其他各种系统发育多样的细菌病原体,在58种合成和测试的设计中。值得注意的是,它们的模型还通过使用已知反应和试剂逐步构建生成的分子来优先考虑生成的分子的合成性。
除了从头产生外,还可以使用深层生成模型进行分子优化(MO)任务。在这种情况下,学习方法旨在通过增强学习(RL)改变现有结构103,104、进化算法3,105,指导搜索在博学的潜在空间中106,或直接转化为具有较高可取性的模拟106,107。这些模型可用于从命中中产生潜在客户,并可以考虑其他相关特性,例如毒性,溶解度,生物可利用性和综合性。
在进入药物发现的后期阶段时,传统的计算化学方法(例如分子动力学(MD))对于理解与目标结合的机制仍然有用。MD以及Markov State Models和自适应抽样等变体可以提供对AI模型选择的见解或用于筛选/排名候选者69,108。相反,DL也可以用来分析Zhu等人的模拟动力学。其中使用卷积VAE和CNN来聚类和比较MD的数据109。
将AI应用于药物发现时仍然存在一些挑战。虽然未标记的化学数据丰富,但高质量标记的数据仍然经常在专有数据库中孤立,或者太难或昂贵而无法收集。多模型模型立即集成了多种数据模式以增强理解,这表明了创建更好模型但需要更大质量的数据集的希望。此外,当前的模型努力产生能够立即满足大量次要特性的候选者,这对于以后的药物开发阶段是必需的。最后,重要的是要了解AI模型的输出以探测其极限并防止幻觉。
结论和未来展望
随着AI技术的进步,它对疾病诊断和药物发现具有巨大的希望,提供了量身定制的干预措施,以打击抗生素效率低下。基于AI的模型分析庞大的临床和实验数据集的能力可以实现精确的疾病诊断,治疗预后以及新型抗菌化合物的预测或发展。这种变革性的潜力扩展到重症监护环境,在该环境中,AI模型增强了诊断准确性并指导个性化治疗策略,最终改善了患者的预后。另一方面,ML和DL正在加速新的抗生素,有助于加强临床前抗生素管道。这是至关重要的,因为传统的药物发现方法滞后,耐药微生物的兴起导致了较不效果的治疗方法。的确,随着AI整合的扩展,其在精炼感染治疗方案和增强抗生素管理方面的作用有望在与AMR的斗争中变得必不可少。
在这种情况下,重要的是要认识到,尽管尖端算法取得了进步,但数据可访问性的局限性仍然带来挑战,从而影响了它们在各种患者人群和临床情况下有效绩效的能力。特别是,必须解决诸如知情同意,数据隐私和算法偏见之类的道德问题。AI模型应使用多样化的代表性数据集开发,以避免永久偏见,并确保所有患者组的公平和准确的结果。实际上,用于AMR诊断,治疗和新的抗菌剂发现的AI模型通常会在不平衡的数据集中训练,这可能会遭受低可靠性的影响。这些模型通常基于固有的偏见数据集,仅捕获来自某些患者组或特定生物学测定的数据。这样的偏见可能反映了人类的认知偏见和数据策划中的社会影响。在这些有偏见的数据集中训练的AI模型可能缺乏概括其训练领域的能力。随着数据策展活动的增长,AI模型将受益于高质量的数据管理和共享协议。确实,当构建数据集时,必须考虑各种参数,包括大小110。此外,尽管大型数据集可以提供详细的见解和支持高级建模,但较小,通知和准确的数据集可能通过避免过多的噪音来提供更多有价值的见解。重点应放在选择与特定目标和设计系统相一致的数据,以便使用自动化和简化的工作流程生成,收集和分析原始数据。例如,由于缺乏专用的公共数据库而阻碍了冶金抗生素的研究,因为它们并未由微笑准确地表示111,112。为了解决这一差距,需要专门的资金和创建强大数据集的努力,这将有助于解锁AI在打击感染性疾病和耐药性方面的全部潜力110。此外,关于AI在AMR中的开发和应用的研究仍在进行中,深度学习算法的黑盒本质可以引起人们对其可信度的关注。
因此,研究人员的数据可访问性显着影响AI模型开发的研究数量和质量,尤其是用于诊断和治疗预测113。当健康数据可用时,研究人员可以构建在广泛的患者人群中更准确和可靠的AI模型。相比之下,对全面和代表性数据集的访问有限限制了强大的算法的开发,使代表性不足的组中的偏见未解决113。为研究人员提供访问多样化的高质量数据对于开发在各个人群中表现良好的AI模型,最小化算法偏见并改善整体结果至关重要。
展望未来,我们预计,在高通量技术和基因组/表达数据库中得出的宽敞,优质数据集的AI模型的利用。这些模型将有助于构建用于快速疾病诊断的系统框架,并提出具有独特作用机理的新型抗生素。对于抗生素发现,这意味着超出了概念概念的命中率证明,还考虑了除毒性和在药物开发后期阶段将AI集成以外的更多二级特性。这些多方面问题的解决方案涉及多个AI模型一起工作,并在此处讨论的所有不同阶段中注入。此外,与医疗保健专业人员,开发人员和监管机构合作对于确保AI负责任地使用至关重要,并持续努力保持公平和透明度。监管指南可能在保护患者数据和促进安全AI部署方面发挥关键作用。
参考
Wong,F。等。利用人工智能对抗传染病。科学 第381章,164â170(2023)。
Cesaro,A。等。深度学习工具加速抗生素发现。专家意见。药物发现。 https://doi.org/10.1080/17460441.2023.2250721(2023)。
Porto,W。F.等。在番石榴抗菌肽的硅硅中,可以为肽设计提供组合探索。纳特。交流。 9,1490(2018)。
刘,G.等人。深度学习引导的抗生素靶向鲍曼尼菌的抗生素。纳特。化学。生物。 19,1342 - 1350,https://doi.org/10.1038/s41589-023-01349-8(2023)。
Stokes,J。M。等。抗生素发现的深度学习方法。细胞 180,688 - 702.E13(2020)。
Naghavi,M。等。全球细菌抗菌抗性的负担,1990年2021年:一种系统分析,预测至2050年。柳叶刀 404,1199年1226年,https://doi.org/10.1016/s0140-6736(24)01867-1(2024)。
Murray,C。J。L.等。2019 年全球细菌抗菌素耐药性负担:系统分析。柳叶刀 第399章,629 - 655(2022)。
De la Fuente-Nunez,C。等。抗生素衰竭:超出抗菌素耐药性。耐药性更新 71,101012(2023)。
Haney,E。F.等。解决抗生素衰竭以外的抗生素衰竭,超出了遗传编码的抗菌抗性。正面。药物发现。 2,(2022)。
Liu,G.-Y.等人。抗菌抗性危机:人工智能可以成为解决方案吗?米尔。Med Res 11,7(2024)。
Ross,J。等。大规模的化学语言表示捕获分子结构和特性。纳特。马赫。英特尔。 4,1256年1264(2022)。
Ferruz,N。等。周年纪念AI的思考。纳特。马赫。英特尔。 6,6 12(2024)。
Safi,Z。等。开发医疗应用程序聊天机器人的技术方面:范围审查。J. Med。Internet Res 22 号,E19127(2020)。
Miner,A。S.等。聊天机器人在与19009年大流行的战斗中。NPJ 数字。医学。 3,65(2020)。
Orsi,M。等。大语言模型可以预测抗菌肽活性和毒性吗?RSC Med。化学。 15,2030年2036年(2024年)。
Smith,K。P.等。人工智能在临床微生物学诊断中的应用。测试。临床。Microbiol Newsl。 42,61 - 70(2020)。
Su,M.-H。等人。使用基于LSTM的多层嵌入老年护理的聊天机器人。在2017年国际橙技术会议(ICOT)70â74(IEEE,2017年)。https://doi.org/10.1109/icot.2017.8336091。Abashev,A。等。
基于Messenger的聊天机器人系统组织的编程工具:对门诊和翻译药物的影响。Bionanoscience 7,403 407(2017)。
Orsi,M。等。使用机器学习来预测钌配合物的抗菌活性**。Angewandte Chemie International Edition 63,E202317901(2024)。
Swanson,K。等。用于设计和验证易于合成且结构新颖的抗生素的生成性AI。纳特。马赫。英特尔。 6,338 A353(2024)。
Wong,F。等。发现具有可解释的深度学习的结构性抗生素类别。自然 https://doi.org/10.1038/s41586-023-06887-8(2023)。
Wan,F。等。通过分子去灭绝发现深度学习的抗生素发现。纳特。生物医学。工程师。 https://doi.org/10.1038/s41551-024-01201-x(2024)。
Wan,F。等。用于抗菌肽识别和设计的机器学习。纳特。比奥恩牧师。 https://doi.org/10.1038/s44222-024-00152-x(2024)。
跳跃者,J.等人。使用 AlphaFold 进行高度准确的蛋白质结构预测。自然 第596章,583年589(2021)。
周,J.等人。通过基于深度学习的序列模型来预测非编码变体的影响。纳特。方法 12,931 - 934(2015)。
Rajkomar,A。等。具有电子健康记录的可扩展和准确的深度学习。NPJ 数字。医学。 1,18(2018)。
新冠肺炎:美国对抗菌抗性的影响,特别报告2022。https://doi.org/10.15620/cdc:117915(2022)。
Hutchings,M。I.等。抗生素:过去,现在和未来。电流。意见。微生物 51,72 - 80(2019)。
Schlander,M。等。研究和开发新药要花多少钱?系统的审查和评估。药物经济学 39,1243年1269(2021)。
Davies,D。了解生物膜对抗菌剂的抗性。纳特。Rev.药物发现。 2,114 - 122(2003)。
什么是败血症? https://www.cdc.gov/sepsis/what-is-sepsis.html(2022)。
Silva,B。N。等。败血症,严重败血症或败血性休克的成年人抗菌治疗的降低。Cochrane 数据库系统。牧师。 2018年,CD007934,https://doi.org/10.1002/14651858.cd007934.pub3(2013)。
Alowais,S。A.等。革新医疗保健:人工智能在临床实践中的作用。BMC 医学。教育。 23,689(2023)。
Rabaan,A。A.等。人工智能在对抗高抗菌抗性率中的应用。抗生素 11,784(2022)。
Van Steenkiste,T。等。使用长期短期记忆神经网络对重症监护病房中血液培养结果的准确预测。阿蒂夫。英特尔。医学。97,38 - 43(2019)。文章
一个 考研一个 谷歌学术一个 Litsa,E。E.等。将质谱转换为De-Novo分子的端到端深度学习框架。
交流。化学。 6,132(2023)。
Smith,K。P.等。通过使用深卷积神经网络对血液培养的自动解释。J.克林。微生物。56,E01521 - 17(2018)。文章
一个 考研一个 考研中心一个 中科院一个 谷歌学术一个 Zagajewski,A。等。大肠杆菌中快速抗菌易感性检测的深度学习和单细胞表型。
交流。生物。 6,1164(2023)。
Mather,C。A。等。通过基质辅助激光解吸电离时间的飞行质谱时间来快速检测万古霉素中间的金黄色葡萄球菌金黄色葡萄球菌。J.克林。微生物。 54,883年890(2016)。
王,H.-Y。等人。基于基质辅助激光解吸电离飞行时间的异质性万古霉素中间葡萄球菌金黄色葡萄球菌的快速检测:使用机器学习方法和无偏见的验证。正面。微生物。 9,2393(2018)。
Weis,C。等。使用机器学习从临床MALDI-TOF质谱进行直接抗菌抗性预测。纳特。医学。 28,164â174(2022)。
Ali,T。等。人工智能用于抗菌抗性预测:实践实施的挑战和机遇。抗生素 12,523(2023)。
Nguyen,M。等。使用机器学习来预测非细类的抗菌MIC和相关的基因组特征沙门氏菌。J.克林。微生物 57,e01260 - 18,https://doi.org/10.1128/jcm.01260-18(2019)。
Teng,Q。等。一项关于医学诊断中深度学习的解释性的调查。多媒体。系统。 28,2335 2355(2022)。
Hakkoum,H。等。医学领域的可解释性:系统的映射和审查研究。应用软计算 117,108391(2022)。
Drouin,A。等。使用无参考基因组比较的预测计算表型和生物标志物发现。BMC基因组学 17 号,754(2016)。
Martãnez-Agã¼ero,S。等。具有智能特征选择的可解释的临床时间序列建模,用于早期预测抗菌多药耐药性。未来一代。电脑系统。 133,68 - 83(2022)。
Lundberg,S。等。解释模型预测的统一方法。ARXIV预印型ARXIV:1705.07874(2017)。
Giacobbe,D。R。等。可解释抗菌管理的可解释的机器学习:机遇和挑战。临床。瑟尔。 46,474 A480(2024)。
Anahtar,M。N。等。机器学习对抗菌抗性问题的应用:转化研究的新兴模型。J.克林。微生物。 59,E0126020(2021)。
Kanjilal,S。等。一种促进门诊抗菌管理的决策算法,用于简单的尿路感染。科学。译。医学。 12,EAAY5067(2020)。
Yelin,I。等。个人临床病史预测了尿路感染的抗生素耐药性。纳特。医学。 25,1143年1152(2019)。
Clark M.等。医疗保健中的聊天机器人:将患者与信息联系起来:新兴健康技术。渥太华(安大略省):加拿大卫生毒品和技术机构(2024)。
Vijayan,A。等。基于自闭症谱系障碍儿童的认知计算的智能学习助理平台的框架。在2018年国际欧洲局控制,沟通和计算会议(IC4)361 365(IEEE,2018)。https://doi.org/10.1109/cetic4.2018.8530940。Morris,R。R.等。
迈向精神卫生应用的人为同理心对话代理:系统设计和用户的看法。J. Med Internet Res 20,E10148(2018)。
Kadariya,D。等。KBOT:知识支持的个性化聊天机器人,用于哮喘自我管理。在2019 IEEE智能计算国际会议(SMARTCOMP)138 A143(IEEE,2019年)。https://doi.org/10.1109/smartcomp.2019.00043。Aljameel,S。S。等。
开发阿拉伯语对话智能辅导系统,用于对ASD儿童的教育。在2017年IEEE计算智能和虚拟环境国际会议的测量系统和应用会议(CIVEMSA)24 29(IEEE,2017年)。https://doi.org/10.1109/civemsa.2017.7995296。Martãnez-Miranda,J。等。
评估用户对基于移动的体现对话剂的可接受性,以预防和检测自杀行为。J. Med Syst。 43,246(2019)。
到目前为止我们做了什么?疾病控制和预防中心,公共卫生数据办公室,监视和技术(2024)。上次审查:2024年4月23日。https://archive.cdc.gov/www_cdc_gov/surveillance/data-modernization/basics/whath-have-have-we-we-done-so-far.html。Naik,N。等。
医疗保健人工智能的法律和道德考虑:谁承担责任?前外科医师。 9,862322(2022)。
Ghanem,M。等。人工智能和个性化医学:改造患者护理。在精密医学的新时代131â142(Elsevier,2024)。https://doi.org/10.1016/b978-0-443-13963-5.00012-1。Bouderhem,R。通过道德和治理塑造AI的未来。
Humanit Soc。科学。交流。 11,416(2024)。
Ueda,D。等。人工智能在医疗保健中的公平性:审查和建议。日本 J. 广播电台。 42,3 15(2024)。
Rehman,A。U.等。人工智能在革新药物发现中的作用。基础研究。 https://doi.org/10.1016/j.fmre.2024.04.021(2024)。
Chenthamarakshan,V。等。通过深层生成基础模型的发现,加速药物靶抑制剂。科学。副词。 9,EADG7865(2023)。
Grisoni,F。等。结合新生药设计的生成人工智能和片上合成。科学。副词。 7,EABG3338(2021)。
Merk,D。等。通过人工智能对生物活性小分子的从头设计。摩尔。信息。 37,1700153(2018)。
Zhavoronkov,A。等。深度学习可以快速鉴定有效的DDR1激酶抑制剂。纳特。生物技术。 37,1038 - 1040(2019)。
Das,P。等。通过深层生成模型和分子动力学模拟加速了抗菌剂发现。纳特。生物医学。工程师。 5,613 - 623(2021)。
Cesaro,A。等人。肽抗生素的高级输送系统。副词。药物Del。Rev. https://doi.org/10.1016/j.addr.2023.114733(2023)。
Miethke,M。等。致力于可持续发现和开发新的抗生素。纳特。化学牧师。 5,726 - 749(2021)。
李,J.等人。膜活性抗菌肽:将机械洞察力转化为设计。神经科学前沿。 11,73(2017)。
Cesaro,A。等。从人血浆中加入蛋白质中的序列的合成抗生素。ACS纳米 16,1880年1895年(2022年)。
Shatri G.等。多性淋赛。Statpearls(2024)。
Nguyen R.等。杆菌肽外用。Statpearls(2024)。
Hansch,C。等。苯氧乙酸与Hammett取代常数和分区系数的生物学活性相关。自然 194,178 - 180(1962)。
黄,J.等人。通过机器学习管道来鉴定有效的抗菌肽,该管道地雷矿化了肽序列的整个空间。纳特。生物医学。工程师。 7,797 A 810(2023)。
Santos-JãºNior,C。D。等。通过机器学习,在全球微生物组中发现抗菌肽。细胞 187,3761â3778.e16,https://doi.org/10.1016/j.cell.2024.05.013(2024)。
Capecchi,A。等。机器学习设计非溶血性抗菌肽。化学。科学。 12,9221 - 9232(2021)。
Torkamannia,A。等。对癌症药物协同预测的机器学习方法的回顾。简短的。生物信息。 https://doi.org/10.1093/bib/bbac075(2022)。
Mayr,A。等。用于药物目标预测Chembl的机器学习方法的大规模比较。化学。科学。 9,5441年5451(2018)。
Bhadra,P。等。AMPEP:使用氨基酸特性和随机森林的分布模式对基于序列的抗菌肽的预测。科学。代表。 8,1697(2018)。
Wu,Z。等。分子:分子机器学习的基准。化学。科学。 9,513â530(2018)。
Krenn,M。等。自我引用嵌入式字符串(自拍照):100%强大的分子字符串表示。马赫。学习科幻。技术。 1,045024(2020)。
Schã¼tt,K。T.等。Schnet - 分子和材料的深度学习结构。J.化学。物理。 148,241722(2018)。
Ma,Y.等人。使用深度学习从人肠道微生物组中鉴定抗菌肽。纳特。生物技术。 40,921â931(2022)。
金,W.等人。使用可解释的子结构生成多目标分子。在第37届机器学习国际会议,ICML 2020Vols Partf168147-7(2020)。
Torres,M。D. T.等。在人蛋白质组中开采加密的肽抗生素。纳特。生物医学。工程师。 6,1451年(2022)。
Torres,M。D. T.等。挖掘人类微生物组揭示了未开发的肽抗生素来源。细胞,按 187,5453 - 5467.E15(2024)。
Maasch,J。R. M. A.等。通过机器学习实现的古代抗微生物肽的分子去渗透。细胞宿主微生物 31,1260 - 1274.e6(2023)。
Cesaro,A。等。AI鉴定的抗生素。纳特。化学。生物。 19,1296年1298(2023)。
Sterling,T。等。锌15的配体发现每个人。J.化学。信息。模型 55,2324年2337(2015)。
贝特曼,A.等人。Uniprot:2023年的通用蛋白质知识库。核酸研究 51,D523âD531(2023)。
Polykovskiy,D。等。分子集(摩西):分子生成模型的基准测试平台。前药。 11,565644(2020)。
金,W.等人。第11章:分子图生成的交界树变异自动编码器。在RSC药物发现系列卷2021年1月(2021年)。
Gã³mez-Bombarelli,R。等。使用数据驱动的分子的连续表示自动化学设计。ACS中心。科学。 4,268 - 276(2018)。
Gebauer,N。W. A.等。针对对称性的3D点集,用于靶向分子的3D点集。在神经信息处理系统的进展32 ARXIV预印Arxiv:1906.00957(2019)。
Jerret R.等。GP-molformer:分子产生的基础模型。(2024)。
Sheth,A。等。将大数据转换为智能数据:通过使用语义和语义网的利用音量,品种和速度来得出价值。IEEE Internet计算3https://api.semanticscholar.org/corpusid:206782839(2007)。
Irwin,R。等。化学物质:用于计算化学的预训练的变压器。马赫。学习科幻。技术。 3,015022(2022)。
Xie,Y。等。火星:多目标药物发现的马尔可夫分子抽样。在ICLR 2021-第9届学习代表国际会议(2021)。
Guimaraes,G。L.等。序列生成模型的客观增强生成对抗网络(器官)。(2017)。
周,Z.等人。通过深厚的增强学习优化分子。科学。代表。 9,10752(2019)。
你,J.等人。目标指导分子图生成的图形卷积策略网络。在神经信息处理系统的进展卷2018年12月(2018年)。
吉田,M。等。使用进化算法和机器学习来探索序列空间以发现抗菌剂。pept。化学。 4,533 - 543(2018)。
Maragakis,P。等。旨在促进药物发现的化学空间的深度学习视图。J.化学。信息。模型 60,4487年4496(2020)。
金,W.等人。学习以分子优化的多模式图翻译。在第七届学习代表国际会议,ICLR 2019(2019)。
Chen,C。H.等。小孔形成抗菌肽的模拟引导的从头设计。J. Am.化学。苏克。 141,4839 - 4848(2019)。
朱,Y.等人。通过深度学习来解密L2-lactamass的共同进化动力学。J.化学。信息。模型 64,3706 3717(2024)。
De la Fuente-Nunez,C。AI在传染病中:数据集的作用。耐药性更新 73,101067(2024)。
Medina-Franco,J。L.等。桥接信息学和药用无机化学:迈向金属果和冶金候选物的数据库。药物发现。今天 27 号,1420 1430(2022)。
Frei,A。等。金属以对抗抗菌耐药性。纳特。化学牧师。 7,202 224(2023)。
阿罗拉,A.等人。基于人工智能的应用程序中对健康数据集的标准值的价值。纳特。医学 29,2929 2938(2023)。
致谢
Cesar de la Fuente-Nunez在宾夕法尼亚大学拥有总统教授职位,是AICHE基金会的Langer奖的获得者,并确认来自IIADR Innovation in Procter&Gamble Company,United Company,United Therapeutics,A A AraL Care奖中的资金BBRF年轻研究员Grant,Nemirovsky奖,Penn Health-Tech Accelerator奖,Dean's Innovation Fund of Perelman医学院的Dean创新基金会宾夕法尼亚大学,美国国立卫生研究院的美国国立普通医学研究所奖励编号R35GM138201和国防威胁减少局(DTRA; HDTRA1-22-10031,HDTRA1,HDTRA1,HDTRA1,HDTRA1-1-1-1-1-1-0014)1-0001)。本文中显示的分子使用Pymol分子图形系统(版本2.5.2Schrã¶Dinger,LLC)渲染。所有数字均在biorender.com中准备。
道德声明
利益竞争
Cesar de la Fuente-Nunez provides consulting services to Invaio Sciences and is a member of the Scientific Advisory Boards of Nowture S.L., Peptidus, and Phare Bio.De la Fuente is also on the Advisory Board of the Peptide Drug Hunting Consortium (PDHC).The de la Fuente Lab has received research funding or in-kind donations from United Therapeutics, Strata Manufacturing PJSC, and Procter & Gamble, none of which were used in support of this work.所有其他作者均声明不存在竞争利益。
附加信息
出版商的注释施普林格·自然对于已出版的地图和机构隶属关系中的管辖权主张保持中立。
权利和权限
开放获取本文根据知识共享署名 4.0 国际许可证获得许可,该许可证允许以任何媒介或格式使用、共享、改编、分发和复制,只要您对原作者和来源给予适当的认可,并提供链接到Creative Commons许可证,并指示是否进行了更改。本文中的图像或其他第三方材料包含在文章的创意共享许可证中,除非在材料的信用额度中另有说明。如果文章的创意共享许可中未包含材料,并且您的预期用途不得由法定法规允许或超过允许的用途,则需要直接从版权所有者那里获得许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/。转载和许可
引用这篇文章
Cesaro, A., Hoffman, S.C., Das, P.
等人。Challenges and applications of artificial intelligence in infectious diseases and antimicrobial resistance.npj Antimicrob Resist3 , 2 (2025). https://doi.org/10.1038/s44259-024-00068-x下载引文
:2024 年 7 月 3 日
:2024 年 11 月 26 日
:2025 年 1 月 7 日
:https://doi.org/10.1038/s44259-024-00068-xhttps://doi.org/10.1038/s44259-024-00068-x