利用电子健康记录增强组学分析的机器学习方法
作者:Aghaeepour, Nima
主要的
组学技术的快速进步彻底改变了生物学的理解。转录组学、代谢组学、蛋白质组学和其他生物分子检测现在可以经济有效地测量单个样品中的大量分析物。尽管这些测定产生高维数据,但临床和预算限制限制了大多数组学研究队列的规模,导致研究结果的重复性较差1,2,3,4。因此,需要创新的分析方法来改进对这些小队列研究的高维数据的分析。
尽管像 Benjamini-Hochberg 程序这样的统计工具可以解决单变量分析中的假阳性率问题,但机器学习的方法较少,其中假阳性表现为过度拟合模型5。最近的一些方法利用了有关哪些特征预计很重要的现有知识,这些知识被用作机器学习模型中特征权重的先验知识6,7。其他方法使用迁移学习,这是一种从预训练数据集中学习机器学习模型的技术,该模型随后用于分析感兴趣的较小数据集8,9。更现代的深度学习方法也已应用于传统的统计框架,例如 Cox 比例风险模型,该模型可用于分析事件时间数据,特别是在经过审查的患者数据集中10。尽管这些方法增强了我们分析高维组学数据的能力,但它们主要侧重于仅从组学数据或信息元数据中学习。在这项工作中,我们利用电子健康记录(EHR)数据来改进组学数据分析。EHR 数据越来越多地通过公共数据集(如 MIMIC11和英国生物银行12)和专有医疗中心数据库(由有意义的使用阶段 2 和观察医疗结果合作伙伴关系 (OMOP) 等标准支持13,14,15)。
现有的多模态机器学习方法通过早期融合(在特征级别)、中间融合(在特定于模态的数据处理后组合潜在表示)或后期融合(结合特定于模态的预测)来组合数据模态16。还有可以混合这些方法的通用框架17 号。然而,获取大量人群的 EHR 数据通常更容易,这给 EHR 组学整合带来了挑战:早期和中期融合通常需要跨模式的完整数据,可能会排除许多患者,而后期融合方法则难以学习跨模式互动18,19。
我们介绍了通过迁移学习增强的临床和组学多模态分析 (COMET),这是一种深度学习架构和迁移学习协议,利用大型观察性 EHR 数据库的迁移学习来改进组学研究中多模态数据集的分析。通过利用预训练,COMET 使用所有可用的 EHR 数据来学习强大的机器学习模型;通过将这些权重转移到多模态架构中,COMET 可以学习跨模态的交互。我们证明 COMET 实现了最先进的预测建模结果,并在两个不同的临床相关任务中实现了更稳健的生物学发现。我们证明 COMET 的 EHR 预训练组件在整个网络中具有正则化效果,提高了模型的性能及其学习广义生物学的能力。我们的工作对于改变我们利用现有 EHR 数据分析组学研究数据的方式具有广泛的影响,并且可以提高我们的发现能力,而无需改变研究设计或增加队列规模。
结果
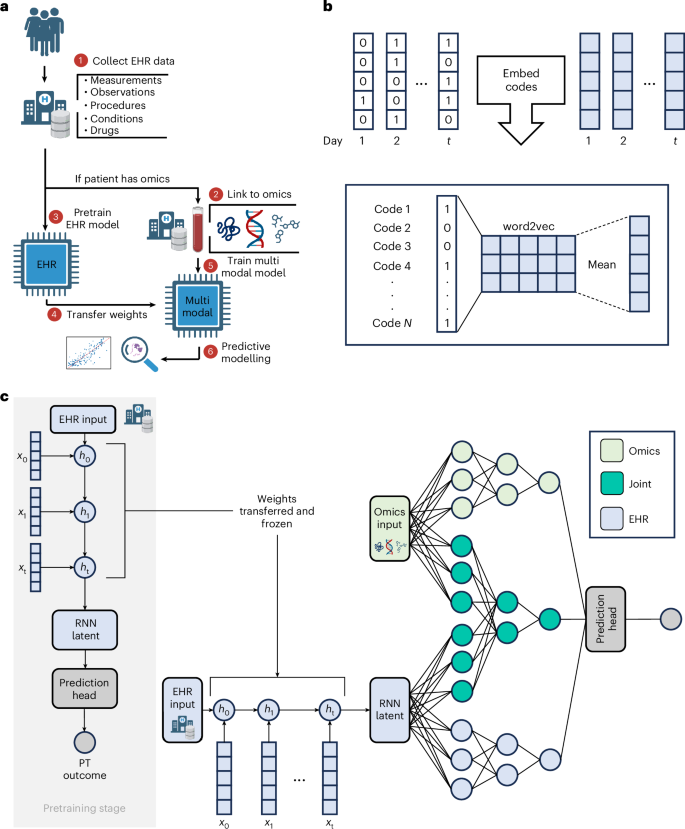
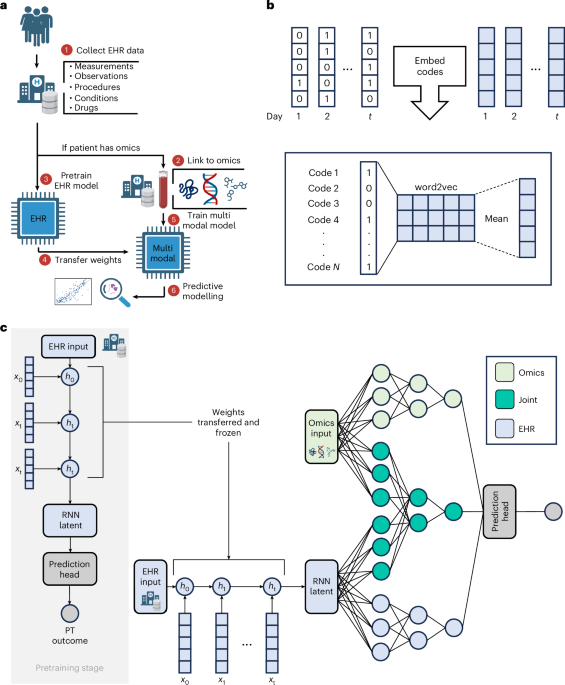
一般来说,当 EHR 数据可用于大量患者并且组学数据可用于较小的子队列时,可以应用 COMET。仅对仅使用 EHR 数据的患者(“预训练队列”)进行训练的模型,将其权重转移到多模态网络,该网络在具有 EHR 和组学数据的较小人群(“组学”)上进行进一步训练和测试。队列™)(图 1)1a)。COMET 由三部分组成: 嵌入纵向 EHR 数据的方法20(如图。1b)、预训练和多模态建模(图 1)1c)。在这里,我们使用 COMET 分析了两个独立队列,一个来自斯坦福大学医疗保健中心的妊娠队列和一个来自英国生物银行的癌症队列。在每个队列中,我们都展示了 COMET 在具有临床意义的预测建模任务方面的最先进性能:分别是临产前的天数或三年全因死亡率。我们使用不同的训练、测试和验证分组执行所有建模实验 25 次,并使用验证集的平均预测来计算性能指标。
COMET 的输入是 EHR 数据和(对于一部分患者)配对的表格组学数据。仅拥有 EHR 数据的患者用于预训练 (PT) 神经网络,仅使用 EHR 数据来预测患者结果。该 EHR 网络的权重被转移到多模态神经网络,用于分析 EHR 和组学数据;神经网络用于预测建模,网络的事后分析用于生物发现。COMET 框架非常灵活,可用于预测任何连续或二元结果。乙,使用 word2vec 将 EHR 数据的 One-hot 编码向量(以白色显示)转换为嵌入(以蓝色显示);对特定一天内发生的每个代码的嵌入进行平均,以计算顺序的汇总嵌入。c,COMET 使用多模态深度学习架构来分析 EHR 数据和组学数据。预训练阶段仅使用EHR数据;核心架构是一个带有门控循环单元的 RNN。预训练后,RNN 权重被冻结并转移到分析 EHR 和组学数据的多模式架构中。控制板一个创建于BioRender.com。COMET 准确预测临产日期
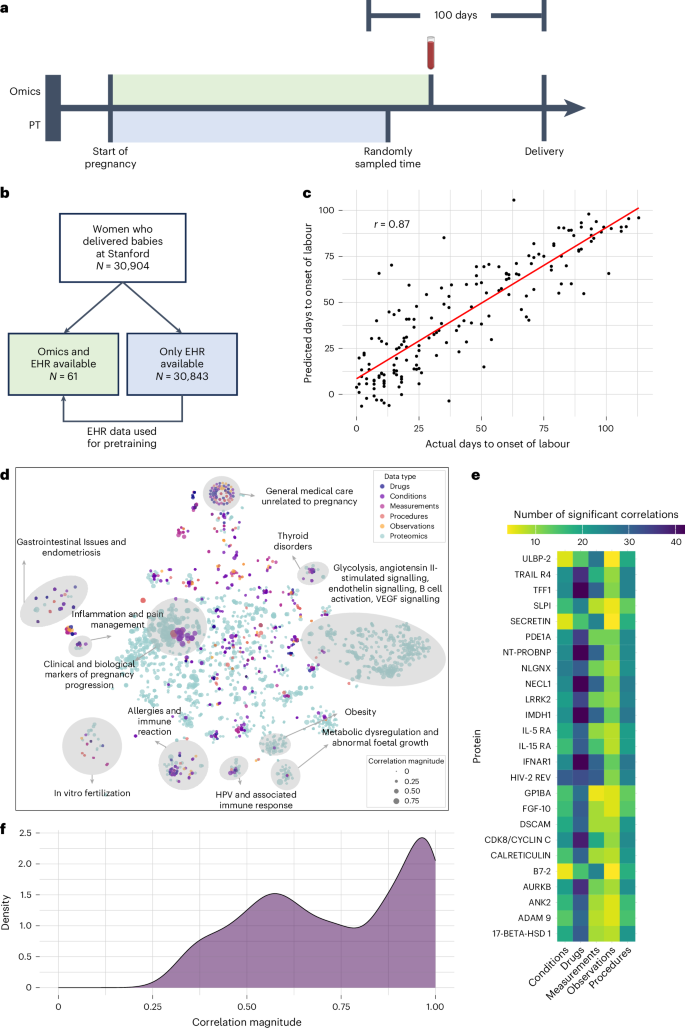
我们首先应用 COMET 来预测孕妇群体中临产的天数(n
2013 年至 2021 年期间在斯坦福大学接生新生儿的 30,904 名患者。所有个体的 EHR 数据均从斯坦福 STARR OMOP 数据库中提取。对于一部分怀孕患者(n–= –61 名患者(组学队列),在怀孕的最后 100 天收集了多个血液(血浆)样本,并用于生成目标蛋白质组数据集,该数据集测量了 1,317 种不同的蛋白质21。我们使用从怀孕开始到血液采样时的 EHR 数据,旨在预测分娩开始的天数(从采样时起)。对于仅有 EHR 数据的患者(n—=—30,843 名患者,预训练队列),没有采样时间(因为这些患者没有蛋白质组学数据)。因此,我们随机选择怀孕最后100天内的一个时间点,使用截至采样时间的EHR数据作为特征,并预测从采样时间到临产的天数作为预训练任务(图1)。2a,b)。图 2:多模式数据揭示了与妊娠进展和临产时间相关的 EHR-蛋白质组学相互作用。一个,对于具有蛋白质组学数据的患者,使用从怀孕开始到蛋白质组学采样时间(绿色阴影)的 EHR 数据构建输入特征;
直到分娩作为我们的预测建模任务的那个时间点)。乙我们利用了在斯坦福大学分娩的女性的数据,并根据她们是否拥有可用的组学数据将她们分为两个群体。c,使用 COMET 框架进行的预测与实际分娩天数进行比较,回归线显示为红色。d,t-SNE 劳动力数据开始的可视化。这些点代表个体特征,并根据模态着色;点的大小基于该特征与分娩开始天数的单变量 Pearson 相关性。仅含有蛋白质变量的簇根据基因本体富集分析进行注释,同时包含临床和蛋白质变量的簇根据临床主题进行注释。e, 热图显示 EHR 特征和蛋白质之间的显着相关性数量(经过 Bonferroni 校正);显示了与 EHR 特征具有最多统计显着相关性的 25 种蛋白质。f, 每个个体 EHR 特征与分娩数据开始时所有蛋白质的最大绝对相关性的分布。我们使用 word2vec 嵌入纵向 EHR 数据,对每天的代码嵌入进行平均。
在使用预训练队列的数据对纯 EHR 架构进行预训练后,我们将权重转移到完整的多模式架构,该架构经过训练以对组学队列进行预测。使用 COMET 预测的预产期与实际预产期之间的 Pearson 相关性很强,这表明 COMET 可以在具有高维数据的小队列中做出高度准确的预测(r≤=≤0.868, 95% 置信区间 (CI) [0.825, 0.900],磷≤=≤3.9≤≤10Ø53,均方根误差(r.m.s.e.)≤=≤16.0)(图 1)2c)。一致性是使用 Lin 的一致性相关系数来衡量的,并在补充表中报告1,这证实了 COMET 的预测与实际的发病时间值非常吻合,没有系统偏差。
我们仅使用 EHR 数据、仅使用蛋白质组数据或两者(“联合基线”)将 COMET 与基线模型进行比较。这些基线仅使用未经预训练的组学队列数据,其架构与 COMET 的相应部分相匹配。仅 EHR 基线仅使用网络的 EHR 部分(图 1)。1c, 浅蓝色)。仅蛋白质组学基线仅使用网络的组学部分(图 1)。1c,浅绿色)。最后,联合基线使用两种数据模式并匹配完整的 COMET 架构。联合基线和 COMET 框架之间的唯一区别是联合基线不包括预训练阶段。仅 EHR 基线表现最差(r≤=≤0.768, 95% CI [0.699, 0.823],磷—=≤1.55≤≤10Ø34,r.m.s.e.-=-20.4-days),并且略优于仅蛋白质组学的基线(r≤=≤0.796, 95% CI [0.733, 0.845],磷≤=≤1.3≤≤10Ø38,r.m.s.e.—=—20.2—天)。联合基线是性能最高的基线(r≤=≤0.815, 95% CI [0.757, 0.860],磷≤=≤7.8≤≤10Ø42,r.m.s.e.—=—18.4—天),但仍不如 COMET。为了确认 COMET 在不同的组学模式中提供益处,我们使用代谢组学对同一队列进行了一组类似的实验,并表明 COMET 的预测建模结果(r�=�0.839, 95% CI: [0.782, 0.881]) 超出了仅根据代谢物预测的性能 (r≤=≤0.758,95% CI:[0.678,0.820])。补充表2列出完整结果。我们还使用岭回归将 COMET 与基线进行了比较,并计算了仅 EHR、仅蛋白质组和联合基线的性能。
为了确定我们是否可以以不同的方式纳入 EHR 预训练,我们使用来自预训练队列的数据训练了仅 EHR 的岭回归模型,并使用受另一项工作启发的岭回归的改编版本7它将预训练模型的系数合并为联合(即多模态)模型中权重的先验。结合预训练可以改善联合基线的皮尔逊相关性(来自r≤=≤0.572, 95% CI: [0.461, 0.665] 至r�=�0.799, 95% CI: [0.737, 0.847]),COMET 仍然优于所有方法(完整结果见补充表3)。最后,我们想要将 COMET 的 word2vec 和基于循环神经网络 (RNN) 的计算 EHR 数据潜在表示的方法与利用转换器的方法进行比较,包括在端到端中学习令牌嵌入方式(我们称之为 COMET Transformer)。
COMET 和 COMET Transformer 的预测之间存在很强的相关性(r≤=≤0.94)。变压器变体的 Pearson 相关性为 0.848(95% CI:[0.800, 0.885]),略低于 COMET(完整结果列于补充表中)4)。总而言之,这些结果证明了无论模型架构如何,纳入预训练的价值,以及 COMET 预测分娩开始天数的卓越能力。COMET EHR™ 蛋白质组学特征相关性分析揭示了对妊娠的生物学见解
COMET 的卓越性能促使我们进一步研究 EHR 和蛋白质组学特征之间的关系,目的是更深入地了解妊娠期间复杂的生物过程。
首先,我们使用了t-分布式随机邻居嵌入(t-SNE)通过将相关矩阵投影到二维来可视化多模态数据;该空间中紧密相连的特征与所有其他变量具有相似的相关性(图 1)。2d)。我们根据每个簇中的 EHR 和/或蛋白质特征所代表的医学概念对这些簇进行了注释。例如,“代谢失调和胎儿生长异常”簇包含代表母亲葡萄糖耐量异常、母亲肥胖和胎儿过度生长的临床代码。它还含有β细胞素和制瘤素M等蛋白质,已知这些蛋白质在葡萄糖稳态和胰岛素敏感性中发挥作用22,23。
同样,我们单独可视化每种 EHR 模态,并使用线条连接显着相关的跨模态变量(补充图 1)。1)。这些可视化结果显示,有许多 EHR 变量与其他特征(包括蛋白质)高度相关,这表明跨模式的信息存在冗余。然而,46.5%的蛋白质与任何EHR特征没有显着相关性,这表明蛋白质组学数据还提供了一些补充信息(补充图1)。2)。
几种蛋白质显示出与 EHR 变量存在大量显着相关性(图 1)。2e),例如干扰素α和β受体亚基1,它与多个感染相关变量相关,与其在免疫功能中的已知作用相一致。为了研究临床数据的附加价值,我们进行了补充分析,计算了每个临床变量与所有蛋白质的相关性,并绘制了最大相关性的分布(图 1)。2f)。这些分析显示了两种模式的重叠和独特信息。COMET 的预训练阶段允许 RNN 提取最有用的信息,并避免包含冗余的、高度相关的 EHR 特征,这可能有助于其与基线模型相比的卓越性能。
COMET 对齐 EHR 和蛋白质组学数据
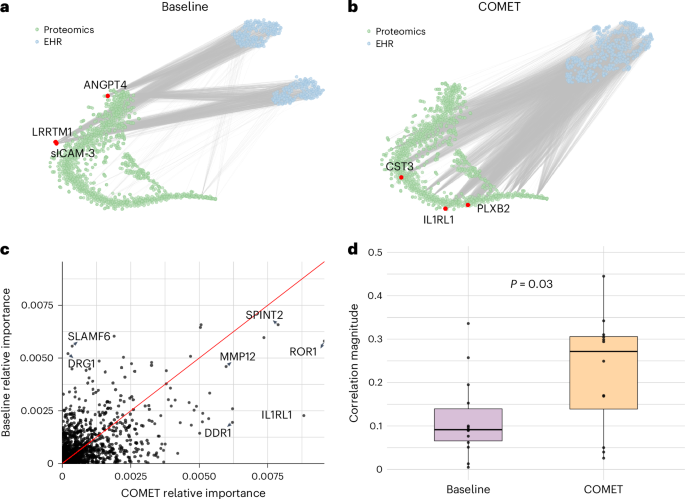
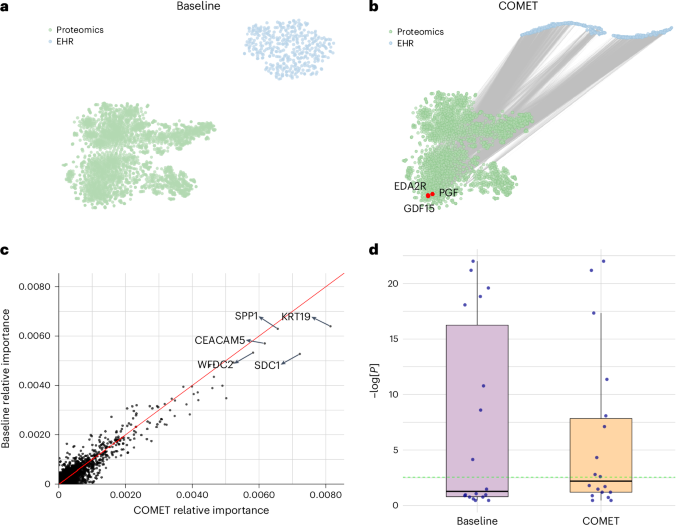
我们通过 EHR 潜在表征检查了 EHR 蛋白质组学关系,可视化 400 个潜在维度中的每一个与每种蛋白质之间的相关性(图 1)。3a,b)。在联合基线模型中学习到的 EHR 潜在表征的维度与所有蛋白质之间存在 3,201 个显着相关性(经过多重假设检验校正)。COMET 的 EHR 潜在表征显示 5,364 个显着相关性,表明 EHR 和蛋白质组数据更好地对齐。一致性的增强表明 COMET 从 EHR 数据中获取的信息更准确地捕捉了患者的潜在生物过程。
,t-联合基线模型中蛋白质组数据和 EHR 潜在表示的 SNE 可视化;线连接统计上显着相关的蛋白质和 EHR 潜在表示的维度。红点代表与 EHR 潜在表示维度具有最多统计显着相关性的三种蛋白质。sICAM-3,可溶性细胞间粘附分子1;LRRTM1,富含亮氨酸的重复跨膜神经元蛋白1;ANGPT4,血管生成素-4;CST3、胱抑素C;PLXB2,丛蛋白-B2;IL1RL1,白细胞介素 1 受体样 1。乙,t-COMET 模型中蛋白质组数据和 EHR 潜在表示的 SNE 可视化。这些线连接了统计上显着相关的蛋白质和 EHR 潜在表示的维度。红点代表与 EHR 潜在表示维度具有最多统计显着相关性的三种蛋白质。c,COMET模型和联合基线模型中蛋白质特征重要性的比较。d,外部数据集中蛋白质丰度与临产天数之间的绝对相关性分布(n基线模型中重要蛋白质的相关性 = = 12nCOMET 模型中重要蛋白质的 = = 14 相关性)。箱线图显示中位数(中心线)、第 25 个和第 75 个百分位数(箱边界),须线延伸到距离箱边缘四分位数范围 1.5 倍以内的最极端数据点。平均绝对相关性之间的差异为 0.109 (95% CI: [0.0134, 0.2052])。t-统计量≤2.39(磷–= –0.0276),估计自由度为 19.1。这种模式在与 EHR 潜在表征维度最相关的蛋白质中最强。
使用 COMET,白细胞介素 1 受体样 1(也称为致瘤性抑制 2 蛋白)、半胱氨酸蛋白酶抑制剂 C 和 plexin-B2 分别与 76%、68% 和 68% 的维度显示出显着相关性。已知这些蛋白质在妊娠进展和分娩时机中发挥作用,并且与之前的研究发现一致24,25,26,27 号,28。EHR 潜在表征与这些蛋白质的高度相关性表明它正在捕获有关患者潜在生物状态的有意义的信息,这可能有助于提高预测建模性能。相比之下,联合基线模型没有表现出这种现象。在基线实验中,与 EHR 潜在表现最相关的蛋白质是可溶性细胞间粘附分子 1、富含亮氨酸的重复跨膜神经元蛋白 1 和血管生成素 4。尽管 angiopoietin-4 确实与妊娠进展存在已知的关联,但其他两种蛋白主要已知与妊娠无关的其他生物学功能,这表明基线模型的 EHR 潜在表现并不能强烈反映潜在的妊娠生物学。补充说明中提供了对这些蛋白质的进一步讨论1。COMET 鉴定出与临产时间相关的蛋白质最后,我们使用积分梯度计算了每种蛋白质的特征重要性,以了解 EHR 潜在表示和蛋白质之间的对齐如何影响模型最终用于进行预测的特征(图 1)。
3c
;完整的特征重要性在补充数据)。众所周知,COMET 模型中具有更大特征重要性的蛋白质与胎龄、胎儿发育或妊娠并发症有关,所有这些都对分娩时间有影响。相反,在联合基线模型中更重要的蛋白质在怀孕中的作用尚不清楚。我们在补充说明中详细阐述了这些蛋白质的已知生物学作用2并通过计算外部数据集中这些蛋白质与临产天数之间的相关性,进一步验证 COMET 模型中重要蛋白质的相关性(图 1)。3d)。COMET 中更重要的蛋白质的平均 Pearson 相关幅度为 0.22 (s.d.=–0.13),而 COMET 中不太重要的蛋白质的平均 Pearson 相关幅度为 0.12 (s.d.=–0.09)。这些分析表明,COMET 不仅通过学习 EHR 数据更具生物学意义的表示来改进预测模型,而且还帮助模型学习准确的生物学。
COMET 改善癌症预后预测
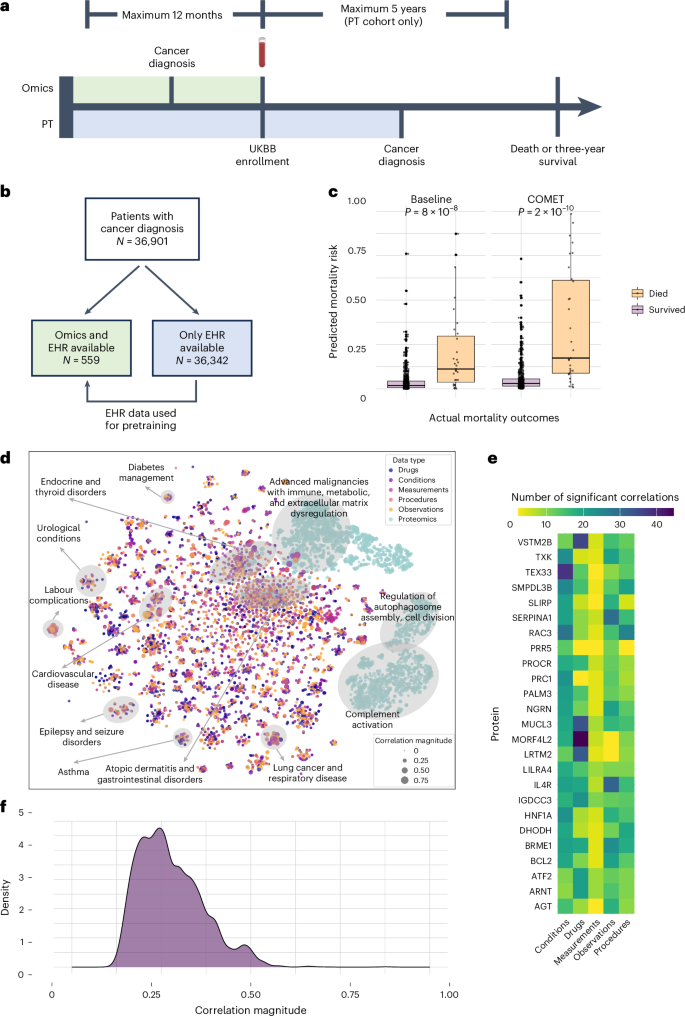
为了展示 COMET 框架的普遍性,我们接下来将其应用于独立群体中的不同预测问题。我们使用 COMET 来预测英国生物银行中癌症患者群体的三年癌症死亡率(n—=—36,901 名患者)11。研究人群包括在英国生物银行注册后 5 年内或最多 12 个月内被诊断为任何类型癌症(通过是否存在以 C 开头的 ICD10 代码确定)的所有患者。这些患者的一部分在参加英国生物银行研究时收集了血液样本,并对这些样本进行了蛋白质组学数据分析29。如果这些患者在初次癌症诊断后 12 个月内收集了样本,我们会将这些患者纳入我们的组学队列(n—=—559 名患者,组学队列)。对于具有蛋白质组数据的患者,我们使用采样时及更早时期的 EHR 数据作为特征;对于其他患者(n–= –36,342 名患者,预训练队列),我们使用了癌症诊断时及更早时期的 EHR 数据(图 1)。4a,b)。图 4:多模式数据提供了有关癌症死亡风险的见解。
,对于具有蛋白质组学数据的患者,我们从采样时间之前的所有 EHR 数据构建输入特征(绿色阴影);对于没有蛋白质组学数据的患者,我们使用 EHR 数据直到癌症诊断(蓝色阴影)。乙我们利用英国生物银行 (UKBB) 中癌症诊断患者的数据,并根据是否有可用的组学数据将人群分为两组。c,COMET 的预测优于表现最好的基线的预测(n–= –559 个预测)。箱线图显示中位数(中心线)、第 25 个和第 75 个百分位数(箱边界),须线延伸到距离箱边缘四分位数范围 1.5 倍以内的最极端数据点。基线预测的平均值差异为 0.089 (95% CI: [0.0469, 0.1277]),双边 Wilcoxon 秩和统计值为 3,431 (磷≤=≤8.37≤≤10Ø8)。COMET 预测的平均值差异为 0.149(95% CI:[0.0892,0.3084]),两侧 Wilcoxon 秩和统计量为 2,537(磷≤=≤1.54≤≤10Ø10)。d,t-SNE 癌症死亡率数据可视化。这些点代表个体特征,并根据模态着色。它们的大小基于与癌症死亡率的单变量相关性。仅包含蛋白质变量的簇根据 GO 富集分析进行注释,同时包含临床和蛋白质变量的簇根据临床主题进行注释。e,热图显示 EHR 特征和所有蛋白质之间的显着相关性数量(经过 Bonferroni 校正)。f,每个 EHR 特征与癌症死亡率数据中所有蛋白质之间的最大绝对相关性的分布。当使用 COMET 通过使用预训练队列来预训练模型的 EHR 部分并将这些权重转移到多模态模型以对组学队列进行预测来预测三年癌症死亡率时,与所有基线(区域)相比,它表现出了优越的性能
受试者工作特征曲线 (AUROC) 下 = = 0.842, 95% CI: [0.744, 0.922],磷≤=≤0,精确召回曲线下面积 (AUPRC) ≤=≤0.504,95% CI:[0.341,0.670],磷≤=≤0;如图。4c)。组学队列中三年死亡率的发生率为 5.5%。基线与临产分析具有相同的设计(有关详细信息,请参阅“COMET 准确预测临产开始天数”部分)。联合基线表现最好(AUROC≤=≤0.786,95% CI:[0.664,0.882],磷≤=≤0,AUPRC≤=≤0.365,95% CI:[0.217,0.555],磷≤=≤0)。仅 EHR(AUROC = 0.749,95% CI:[0.636,0.843],磷≤=≤0,AUPRC≤=≤0.205,95% CI:[0.122,0.349],磷�=�0) 和仅蛋白质组学 (AUROC�=�0.737, 95% CI: [0.634, 0.838],磷≤=≤0,AUPRC≤=≤0.325,95% CI:[0.179,0.495],磷–= –0) 基线也显示了一些预测建模信号。一致性使用 Cohen’s kappa 进行测量,并在补充表中报告5,展示了超出所有基线的一致且可靠的分类性能。与分娩实验的开始一样,我们将 COMET 的性能与逻辑回归基线进行了比较,包括结合了先验知识的适应,同样显示出在结合预训练模型的先验知识时,基线 AUPRC 的优势为 0.263–0.279。
完整结果列于补充表中6,这表明 COMET 超过了所有逻辑回归基线,包括结合了预训练先验的适应。我们还使用 COMET Transformer 进行了实验,这再次表明预测之间存在很强的相关性(r�=�0.72),COMET 优于 COMET Transformer(补充表7)。无论模型架构如何,当包含预训练时,预测建模性能都会得到提高,并且 COMET 的性能超过所有其他方法。
多模态数据揭示癌症预后的生物学
我们用过t-SNE 可视化跨模态的所有变量对之间的相关矩阵,以更好地理解它们的关系(图 1)。4天)。与劳动力数据的开始相比,蛋白质组学数据和电子病历数据模式之间的重叠较少。然而,当可视化每个模态的相关网络时,我们确实看到蛋白质组数据和 EHR 数据模态之间存在显着相关性(补充图 1)。3)。
为了深入了解这一现象,我们计算了每个蛋白质变量与所有 EHR 变量的显着相关性数量(图 1)。4e)。在所有蛋白质中,死亡因子 4 样蛋白 2 与 EHR 变量(尤其是药物处方)的相关性最多。死亡因子 4 样蛋白 2 与肿瘤动力学和治疗反应相关,这可能解释了它与药物订单的高度相关性30。我们发现癌症患者中很大一部分蛋白质(65.9%)与其任何 EHR 变量都没有显着相关性(补充图 1)。4)。我们计算了每个 EHR 特征与所有蛋白质的相关性,并计算了每个 EHR 特征的所有蛋白质的最大相关性(图 1)。4f)并发现许多 EHR 特征与癌症患者的所有蛋白质相关性较低。这一发现重申了在我们的分析中包含多种数据模式的价值。当研究 EHR 特征和蛋白质之间的强相关性时,它使我们能够发现跨数据模式的有趣关系。例如,慢性 B 细胞淋巴细胞白血病的诊断与淋巴细胞激活基因 3 蛋白强度的相关性最高(r≤=≤0.46, 95% CI: [0.333, 0.571],磷≤=≤8.4≤≤10—31);淋巴细胞激活基因3是一种在白血病细胞上表达的免疫检查点,已被证明是一种有效的预后标志物(补充图1)。5)31。
COMET EHR 表述反映了已知的癌症生物学
我们再次可视化 EHR 潜在表示和蛋白质组数据之间的关系(图 1)。5a、b)。在联合基线实验中学习到的 EHR 潜在表示的维度与任何蛋白质都没有显着相关性,而 COMET 的 EHR 潜在表示的维度有 7,591 个统计上显着的相关性,表明这种对齐效应发生在整个数据集上。与 COMET EHR 潜在表征具有最多显着相关性的所有蛋白质均已被证明是癌症的预后生物标志物。我们在补充说明中详细阐述了这些蛋白质3。这些发现表明,COMET 不仅有效地对齐了 EHR 和蛋白质数据,而且还揭示了与已知癌症预后标记物一致的具有生物学意义的相关性,强调了这种方法在跨不同数据集识别临床相关生物标记物和治疗靶点方面的潜力。
-联合基线模型中蛋白质组数据和 EHR 潜在表示的 SNE 可视化。这些线连接了统计上显着相关的蛋白质和 EHR 潜在表示的维度。红点代表与 EHR 潜在表示的维度具有最多统计显着相关性的三种蛋白质。乙,t-COMET 模型中蛋白质组数据和 EHR 潜在表示的 SNE 可视化。这些线连接了统计上显着相关的蛋白质和 EHR 潜在表示的维度。c,COMET模型和联合基线模型中蛋白质特征重要性的比较。d, 单变量分布磷值(从t-测试)根据外部数据集中的三年死亡率比较蛋白质水平(n☀=☀18磷基线模型中重要蛋白质的值和n☀=☀18磷COMET 模型中重要蛋白质的值)。绿色虚线代表 Bonferroni 调整的显着性阈值。
COMET 模型验证了已建立的癌症预后标志物
COMET 模型中具有较高特征重要性的蛋白质与已知的预后生物标志物一致(图 1)。5c,完整的特征重要性在补充数据)。我们在补充说明中详细阐述了这些蛋白质4。我们进一步验证了 COMET 模型中更重要的蛋白质比基线模型中更重要的蛋白质与死亡率的相关性更高(图 1)。5天)。我们发现 COMET 模型中最重要的 18 个匹配蛋白中有 9 个在统计上与死亡率状态显着相关,而只有 8 个来自联合基线模型。此外,中位数磷COMET 蛋白的值较低。这些发现进一步验证了彗星模型可以更好地与已知生物学保持一致。
彗星通过初始化充当正规化的形式
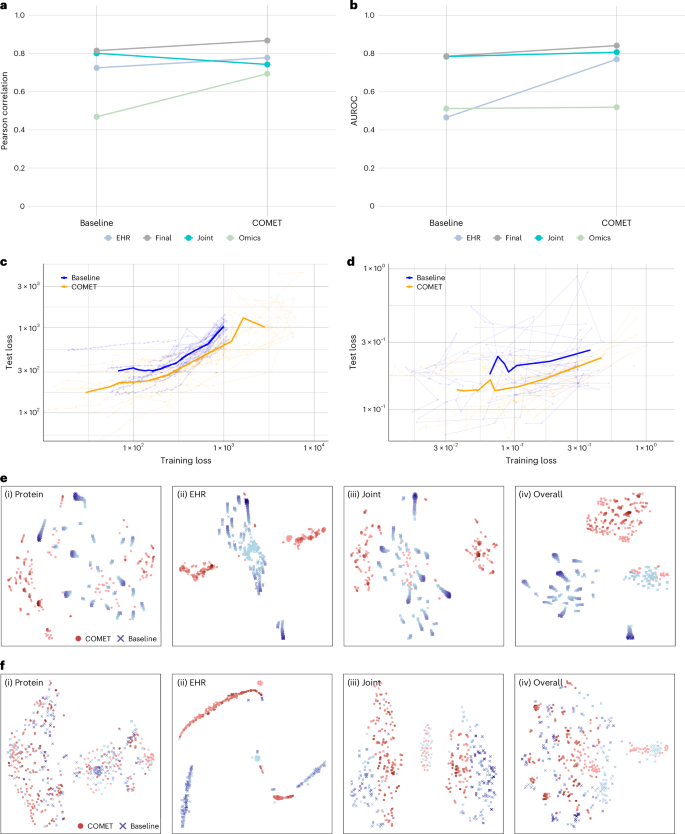
为了更好地了解网络的哪一部分负责预测建模的改进,我们研究了网络倒数第二层中中间节点的性能(图。6a,b)。正如预期的那样,我们看到了与彗星的EHR节点的改进,这大概是由于用于预处理模型的其他EHR数据。上面讨论的生物学表示的改进还表明,蛋白质组学和/或关节节点也可能有所改善。实际上,我们看到了这种效果(从劳动分析开始中的蛋白质组学节点以及癌症分析中的关节节点)。这些发现支持以下假设:彗星不仅可以提高模型从EHR数据中学习的能力,还可以从OMICS数据中学习。我们还表明,网络的OMIC和联合部分中的权重是这些转移权重的函数(补充说明5);因此,理论上也支持这样的发现。
,与彗星相比,每个中间节点上的值与几天与劳动的发作之间的Pearson相关性。乙,与彗星相比,每个中间节点上的值的AUROC用于预测联合基线模型中的三年死亡率。c,每个时期劳动实验发作的每次迭代的训练损失与测试损失,将关节基线与彗星进行比较;平均损失以粗体显示。d,每个时期癌症死亡率实验的训练损失与测试损失,将关节基线与彗星进行比较;平均损失以粗体显示。e,可视化关节基线和彗星模型的参数空间,以预测劳动发作的天数;每个点表示训练期间一个时期的参数。较早的时期以较浅的颜色显示。蛋白质参数空间(I),EHR参数空间(II),关节参数空间(III)和总参数空间(IV)。f,关节基线和彗星模型的参数可视化以预测癌症死亡率。每个点代表训练期间一个时期的参数空间。较早的时期以较浅的颜色显示。蛋白质参数空间(I),EHR参数空间(II),关节参数空间(III)和总参数空间(IV)。
为了了解这种改进的机制,我们将训练损失与彗星模型与基线模型之间的测试损失进行了比较(图。6c,d)。我们观察到使用彗星时任何给定的训练损失的测试损失均较低,这表明彗星可以通过充当正则化形式来提高概括性。我们探索了这种正则化效果如何影响实际模型参数。
通过可视化参数空间(方法),我们可以看到,与基线模型相比,彗星模型占据了参数空间的单独部分(图。6e,f)。这表明正则化效应允许参数收敛到参数空间的一部分,该空间会导致更具概括性和更精确的模型。在整个训练中,通过参数空间的25个迭代中的每一个路径都在补充无花果中可视化。613。总之,由于模型能够从EHR和OMICS数据中学习的能力,这是由正则化效应启用的,这是由于模型从EHR和OMICS数据中学习的能力,这是由于彗星在RNN中的初始化而产生的。转移学习。
讨论
我们证明了彗星通过预处理和转移学习来改善各种任务的预测建模的能力,从而使参数空间的先前无法达到的部分访问。彗星会产生更好的正则模型,从而更准确地反映已知的生物学并鼓励EHR OMICS对准。重要的是,我们表明,使用EHR预审进可以改善不同深度学习体系结构的预测建模性能以及脊回归和逻辑回归基准。通过将EHR与OMIC数据集成,彗星先驱者在生物医学中的多模式分析,超越了传统的单模式方法19,32,33,34,35。据我们所知,彗星是利用EHR转移学习来改善OMICS数据分析的第一种方法。
彗星通过RNN预处理的权重对关节和OMICS网络组件中的梯度计算的影响通过反向传播来改善生物建模。因此,彗星模型确定了针对特定健康结果的生物学相关蛋白质。在劳动模型的开始中,它突出了对免疫调节,胎盘发育和妊娠并发症至关重要的蛋白质(白介素-1受体样1,胱抑素C,SPINT2,DDR1,VEGFR SR3和MMP12)。对于癌症死亡率,它鉴定出参与肿瘤增殖和微环境调节的蛋白质(CECAM5,KRT19和SDC1),表明彗星揭示有意义的分子机制的能力。
这项研究有几个局限性。尽管我们已经证明可以将彗星应用于回归和分类任务,但仍需要进一步的研究来确定它是否可以推广到不同的体系结构。对于某些OMICS模式(例如,空间转录组学)所需的必要条件,如果OMICS架构更为复杂,彗星是否会导致类似的改进是未知的。此外,EHR数据是OMOP提取物,它们是手动映射并可能包含错误的。未来的工作将集中于评估对其他数据结构和体系结构的概括性。最后,彗星框架需要在EHR预处理数据集中的未来工作中标签,将探索自我监管的预读任务,尤其是考虑到最近的工作表明EHR基础模型可以直接预测蛋白质表达水平36。
COMET 通过利用 EHR 数据增强组学分析、改进预测模型和生物发现,推进多模式生物医学数据集成。它超越了简单的病例对照分类,捕捉到了可能被还原论研究设计所掩盖的微妙疾病状态。其正则化特性提高了模型的通用性和鲁棒性,增强了潜在的临床转化。随着多模式生物医学数据可用性的增长,COMET 为阐明临床表型和分子机制之间的复杂关系提供了基础,并可以改变我们分析组学研究数据的方式。
方法
数据集
研究中使用的两个队列来自真实世界的临床研究。第一个数据集包括在怀孕的最后100天内从斯坦福的61名妇女收集的系列血液样本20。所有妊娠均出现自然临产(即排除剖腹产和药物引产病例)。在补充表中描述了患者的人口统计8。每个患者收集了两到三个样本。分析中总共使用了 171 个样本。训练/测试/验证的划分发生在患者层面;因此,来自患者的所有样本都包含在同一个数据分割中。使用基于适体的平台分析血浆样本以测量 1,317 种蛋白质。我们排除了 12 种用作对照的蛋白质;因此,分析仅考虑了 1,305 种蛋白质。将研究数据与 EHR 记录关联起来已得到机构审查委员会的批准(斯坦福 IRB #39225)。
英国生物库是一个大型生物库37。一部分参与者提供了使用基于适体的平台分析的血液样本,以测量来自53,058名参与者的2,894蛋白的中位数28。参与者的人口统计在补充表中描述9。我们仅使用英国生物银行研究注册时收集的血液样本生成的蛋白质组学数据;因此,我们分析中的每位患者都有一份血液样本。
EHR 数据和预训练队列的提取
两个数据集的EHR数据均以OMOP格式提供14。对于我们数据集中的患者,我们从测量、观察、药物暴露、条件发生和程序发生表中提取所有数据。我们删除 Concept_id 为 0 的行。
对于劳动数据集开始时的OMICS队列,我们从怀孕开始(定义为分娩前的280天)才得出数据,直到OMICS取样为止(图。2a)。对于劳动数据集开始时训练的队列,我们模拟了原始OMICS研究的设计,在该研究中,我们在分娩和最多100天之间进行了随机日期。然后,我们使用该日期及之前的 EHR 数据,直至怀孕开始。
对于癌症死亡率数据集中的OMICS队列,我们从蛋白质组学抽样和更早的时候提取EHR数据(图。4a)。对于癌症死亡率数据集中的训练列表,我们根据第一次出现任何ICD10代码来计算癌症诊断的时间,该代码以C开头。我们从癌症诊断和更早的时候提取数据。我们使用OMOP中的死亡表来识别死亡率。
嵌入过程
劳动发作
为了使EHR数据在深度学习模型中更适合分析,我们首先学习每个代码的嵌入式。为此,我们在上述EHR数据表中提取独特的概念代码。然后,我们按患者和日常对代码进行分组。在特定日期内为患者发生的所有代码都被视为句子中的单词。它们被随机改组,因为许多EHR变量(尤其是条件)的特定时间戳是不可靠的。然后,我们使用word2vec学习这些句子的每个词(概念代码)的400维嵌入(在特定日内代表所有临床事件的代码顺序)38。为了训练和奥理学训练,学习了单独的Word2Vec模型。学习嵌入后,我们将嵌入的平均值用于特定日期内发生的代码。现在,我们在特定一天内为所有患者提供了一个摘要嵌入所有EHR数据。这些摘要嵌入的序列将依次馈入RNN进行预测建模。从最近的日期开始,我们最多使用32天的数据。
癌症死亡率
我们使用与上述相同的过程。
彗星深度学习体系结构
实验的体系结构略有不同,因为一个是一个回归任务,另一个是分类任务。通常,这两个架构都有一个EHR组件,一个关节组件和一个组分。
劳动发作
网络的EHR组件由带有封闭的复发单元的RNN组成,然后是单个线性层,该线性层采用了最后一个RNN层的输出,并生成了基于EHR的单个预测。选择层,隐藏尺寸和辍学的数量是超参数。该体系结构用于仅EHR基线。网络的OMICS组件是单个线性层,该线性层将OMIC作为输入并生成基于OMICS的单个预测。该部分的体系结构用于仅OMIC的基线。关节层是一个单个线性层,该层采用串联的EHR潜在表示和OMICS数据,并生成一个单个关节预测。网络的最后一层是单个线性层,没有偏置,将三个预测结合到最终预测中。通过最大程度地减少平方误差损失来训练网络:
$$ {{\ rm {MSE}}}} = - \ frac {1} {n} {n} \ Mathop {\ sigma} \ limits_ {i = 1}^{n}^{n} {n} \ left(\ rm {true}}}}} \ log \ left [{y} _ {{{\ rm {\ rm {pread}}}}}}}} \ right]+\ left(1- {y} _ {y} _ {{{{\ rm {true}}}}}}}}}}} \ right)\ log\ left [1- {y} _ {{{\ rm {pread}}}}}}} \ right] \ right)。$$
癌症死亡率
癌症死亡率数据集的架构与上述相似,因为该模型用于分类而不是回归,因此具有较小的变化。我们使用一个具有一个隐藏层的多层感知器,并在输入和隐藏层之间具有整流的线性单位激活功能,并在网络末端和模型的仅OMICS的末端包括一个Sigmoid。我们略微改变了体系结构,以证明彗星不是特定于建筑的,并且在多个深度学习架构设计中,该框架可能是有益的。通过最大程度地减少二进制跨透明拷贝损失来训练网络。
$$ {{\ rm {bce}}}} = \ frac {1} {n} \ Mathop {\ sigma} \ limits_ {i = 1}^{n} {n} {(\; {y} _}{true}}}}} - {y} _ {{{\ rm {pread}}}}}})}}^{2} $$
彗星高参数细节
为了确定超参数,我们使用三倍的交叉验证和网格搜索。在两个培训折叠中,我们将数据的20%作为早期停止的测试集。我们通过网格搜索在验证集上评估每个高参数集的性能,并选择在三倍交叉验证范围内验证集的最低平均损失的高参数集。这些超参数用于所有随后的实验,包括具有不同火车,测试和验证分裂的实验。对于网络的EHR部分,参数网格如下:Learning_rate,{1 - 101,1 - 102,1 - 103,1 - 104};辍学,{0.1、0.2、0.3、0.4、0.5};lr_decay,{1 - 101,1 - 102,1 - 103,1 - 104};图层,{2,4};hidden_dim,{400}。批次尺寸固定为512,用于训练训练的队列,其中Omics队列为16。对于仅蛋白质组学实验,我们从相同范围内分别优化了学习率和LR_DECAY。从联合实验中,我们优化了学习率,辍学和LR_DECAY,但将图层和隐藏尺寸固定为从仅EHR实验中选择的最佳权重。当我们转移权重时,我们将层和辍学的数量固定为预处理实验的最佳值。在癌症实验中,我们进一步优化了网络蛋白质组学部分中的隐藏尺寸(从值16、32和64),因为它包括两层。两组实验的最佳超参数显示在补充表中10和11。
使用这些超参数,我们使用不同的列车,测试和验证拆分进行了25个实验的迭代。培训集是数据的70%,测试和验证集为15%。如果至少连续五个时期的测试组中的损失没有减少,我们就进行了尽早停止。为了计算最终性能指标,我们将所有25个迭代中的验证设置的预测平均,并使用这些平均预测来计算最终预测。
基于变压器的体系结构
我们修改了彗星,以利用基于变压器的体系结构来学习和计算EHR数据的潜在表示,以代替Word2Vec嵌入代币并使用RNN。如上一节所述,我们维护了其余的彗星体系结构,以集成OMICS数据。
所有EHR数据都将预处理为顺序令牌。我们在序列的开头以及标记任何给定日期的开始和结束时都包括特殊令牌。最大序列长度为1,024,6.8%的患者的序列序列更长,而其最古老的EHR数据被排除在外。网络首先将每个临床代码映射到学习的128维嵌入向量。这些嵌入按d缩放,以在随后的操作过程中保留幅度。为了编码时间信息,我们结合了正弦的位置编码PE(POS,2我)= sin(pos/10,000(2我/d))和PE(POS,2我+1)= cos(pos/10,000(2我/d))位置POS和维度我。这些编码被添加到缩放的嵌入中,以产生位置感知的输入表示。核心变压器使用两个编码器层,每个层都使用四个头的多头自我注意力。遵循注意力机制,有一个两层馈电网络。
第一层将中间表示形式投射到512维空间中,应用了一个整流的线性单位非线性,然后辍学,然后将其投射回128个维度。为了处理可变长度序列,我们实施了注意力掩模,其中填充了填充令牌是较大的负注意分数,从而有效地将它们排除在注意计算之外。最后一个位置的最终隐藏状态是潜在的患者表示形式,其使用方式与网络下游部分中的RNN潜在表示相同。
基于变压器的体系结构详细信息
为了确定超参数,我们使用三倍的交叉验证和网格搜索。在两个训练折叠中,我们将数据的20%作为验证设置,用于提早停止。我们通过网格搜索在验证集上评估每个高参数集的性能,并选择在三倍交叉验证范围内验证集的最低平均损失的高参数集。这些超参数用于所有随后的实验,包括具有不同列车,测试和验证拆分的实验。为了预处理网络的EHR部分,参数网格如下:Learning_rate,{1 - 102,1 - 103};辍学,{0.1,0.3};lr_decay,{1 - 102,1 - 103};此外,层的数量固定为2;隐藏尺寸,固定在128;批次尺寸,固定在64。对于基线OMICS实验,我们使用{1的网格Learning_rate 102,1 - 103,1 - 104},{0.1、0.2、0.3、0.4、0.5}的辍学和{1的lr_decay 102,1 - 103,1 - 104};层数固定为2;隐藏尺寸,固定在128;和批处理大小,固定为16。
当我们转移权重时,我们将辍学固定为预训练实验中使用的最佳值。在癌症实验中,由于硬件的不同硬件,预处理的批量大小为32,因为在英国生物银行的研究分析平台上进行了这些实验。我们不会重新运行OMICS实验,因为架构中唯一的变化是分析EHR数据的网络部分。补充表中显示了劳动实验发作的最佳超参数12对于癌症死亡率实验,在补充表中13。
使用这些超参数,我们使用不同的列车,测试和验证拆分进行了25个实验的迭代。培训集是数据的70%,测试和验证集为15%。如果至少连续五个时期的测试组中的损失没有减少,我们就进行了尽早停止。为了计算最终性能指标,我们将所有25个迭代中的验证集中的预测进行了平均,并使用这些平均预测来计算最终预测。
脊回归基线
我们实施了脊回归基线,包括适应,可以在预处理中得出的系数上纳入先验。通过消除低变异功能(阈值= 0.01)并应用标准归一化来预处理所有功能。EHR功能是单热编码的(也就是说,如果发生代码,则功能值为1;否则为0)。对于模型选择,我们使用患者ID进行了三倍的交叉验证,以防止相关样本之间的数据泄漏。高参数网格包括我”{0.1,1,1,5,10,25,50,75,100,250,500,1,000},对于结合先前的先验优势的模型γ{0,0.25,0.5,0.75,1}。该模型最大程度地减少了目标||y -Xî²||2+我”|| - -。0||2, 在哪里n是样本大小, -0表示从预处理和γ控制这些先验的影响。如果该功能不在验证的数据中(即所有蛋白质组学特征),则 -0设置为0。根据最低平均R.M.S.E.选择最佳的超参数。跨验证褶皱,并在补充表中显示14。为了进行最终评估,使用交叉验证的固定样品生成预测,以确保绩效估计无偏见。
逻辑回归基线
我们实施了逻辑回归基线,包括可以在预处理中得出的系数上纳入先验的改编。通过消除低变异功能(阈值= 0.01)并应用标准归一化来预处理所有功能。EHR功能是单热编码的(也就是说,如果发生代码,则功能值为1;否则为0)。对于模型选择,我们使用随机拆分的三倍交叉验证。超参数网格包括正则强度C{10â»8,10â»7,10â»6,10â»5,10â»4,10â»3,10â»2}和用于纳入先验的模型,先前的权重γ{0,0.25,0.5,0.75,1}。该模型最大程度地降低了目标的数量L]+â(1/2C)|| - -。0||2, 在哪里L是标准的逻辑似然函数:\( - {\ rm {log}}}({\ prod} _ {i} \,{p}({x__ {i}})({1-p({x} _ {i})})^{({1-y} _ {i})})\)\)\)。这里p(x)= 1/(1+ - e(X)), -0表示从预处理和γ控制这些先验的影响。使用牛顿拉夫森更新优化了参数。根据验证折叠的最大平均AUROC选择最佳的超参数,并在补充表中显示15。为了进行最终评估,使用交叉验证的固定样品生成预测,以确保绩效估计无偏见。
外部验证
劳动发作
我们使用了由孕妇的血浆蛋白质组学组成的外部数据集来确定在彗星模型中鉴定为更重要的蛋白质是否与其他患者队列的劳动开始更加密切相关39。我们考虑了在彗星模型中更重要的50种最重要的蛋白质(在50个中,外部数据集中有14种蛋白质),并计算了这些蛋白质的Pearson相关性,几天才能发作。我们将其与在基线模型中更重要的50种最重要蛋白的Pearson相关性(在50个中,外部数据集中有12种蛋白质)。
癌症死亡率
我们使用了由乳腺癌患者的蛋白质组学组成的外部数据集来确定在彗星模型中鉴定为更重要的蛋白质是否与其他患者同类中的癌症死亡率更密切相关40。我们考虑了在彗星模型中更重要的50种最重要的蛋白质(在50个中,外部数据集中有18种蛋白质),并计算了这些蛋白质的Pearson相关性,以达到劳动发作。我们将其与在基线模型中更重要的50种最重要蛋白质的Pearson相关性(在50个中,外部数据集中有18种蛋白质)。
中间节点预测
对于依赖于网络EHR,蛋白质和关节部分中间表示的每个分析,我们在预测头之前使用节点。这些由浅绿色(OMIC),浅蓝色(EHR)和蓝绿色(关节)节点表示,这些节点将直接馈入预测头(图。1c)。这些节点中的值仅是OMIC数据的函数,仅是EHR数据和两个数据模式。
参数空间可视化
很难可视化复杂的神经网络的所有参数,并且可能没有意义,因为具有不同参数的网络在功能上可能是相同的。因此,就像其他人所做的那样,我们比较每个网络表示的函数,而不是比较参数7,41,42。对于每个模型(在每个时期),我们输入所有数据点,计算输出并将输出连接到单个向量(包括上述中等节点预测的值,用于可视化蛋白质,EHR和EHR和关节参数空间)。最终输出用于可视化整体参数空间。这些向量被串联成单个矩阵,该矩阵使用t-sne。报告摘要
有关研究设计的更多信息,请参阅
自然投资组合报告摘要链接到这篇文章。数据可用性
妊娠队列的蛋白质组学数据可在Dryad获得(
http://datadryad.org/和https://doi.org/10.5061/dryad.280GB5MPD)。由于斯坦福大学的政策,怀孕队列的电子病历数据无法公开共享。癌症死亡率队列的数据(蛋白质组学和EHR)可通过英国生物库获得,但由于英国生物银行的数据使用政策,无法公开共享。提取我们研究中使用的队列的查询包括在《代码》中https://github.com/samson920/comet/tree/main,并批准获得英国生物库的研究人员可以使用这些笔记本和GitHub教程来复制我们的分析。用于外部验证劳动特征重要性的数据集可用https://nalab.stanford.edu/multiomicsmulticohortpreterm/。用于外部验证癌症死亡率特征重要性的数据集可在参考文献的补充数据中找到。40。
代码可用性
所有代码都可以在https://github.com/samson920/comet/tree/main并通过Zenodo(https://doi.org/10.5281/Zenodo.13977341)43。所有Stanford Machine Learning算法均在Nvidia Tesla P40,24 gb GPU VRAM上进行了培训。所有英国生物银行机器学习模型均在英国生物银行研究分析平台的 NVIDIA T4 GPU 上进行训练。
参考
Schã¼ssler-Fiorenza Rose,S。M.等。用于精准健康的纵向大数据方法。纳特。医学。 25,792 - 804(2019)。
Karczewski,K。J.&Snyder,M。P.健康与疾病的综合幻影。纳特。吉内特牧师。 19,299 - 310(2018)。
基尔皮奇,A.等人。组学数据中的变量选择:小样本量的实际评估。公共科学图书馆一号 13,E0197910(2018)。
珀恩(W.代谢物 10,286(2020)。
Benjamini, Y. & Hochberg, Y. 控制错误发现率:一种实用而强大的多重测试方法。J.R.统计。苏克。乙 57,289 300(1995)。
Ruiz,C.,Ren,H.,Huang,K。&Leskovec,J。具有辅助知识图的高维,深度学习。副词。神经信息。过程。系统。 36,26348 26371(Curran Associates,2023)。
Culos,A。等。将机械免疫学知识的整合到机器学习管道中可以改善预测。纳特。马赫。英特尔。 2,619 - 628(2020)。
Jiang,Y.,Alford,K.,Ketchum,F.,Tong,L。&Wang,M。D. TLSURV:通过多阶段转移学习整合多矩数据,以进行癌症存活预测。在过程。第11届ACM生物信息学,计算生物学和健康信息学国际会议23(ACM,2020)。
Rong,Z.中文电子。 30,843 - 852(2021)。
Katzman,J。L.等。DeepSurv:使用COX比例危害深神经网络的个性化治疗建议系统。BMC 医学。资源。methodol。18,24(2016)。Goldberger,A。L。等。
Physiobank,PhysiotoolKit和Physionet:用于复杂生理信号的新研究资源的组成部分。循环 101,E215 e220(2000)。
Papez,V。等。将英国生物库转换为Covid-19研究及其他地区的OMOP共同数据模型。J. Am.医学。通知。副教授。 30,103 - 111(2022)。
DâAmore,J。D。等。有意义的使用第2阶段认证的EHR是否可以互操作性?Smart C-CDA合作的发现。J. Am.医学。通知。副教授。 21,1060年1068(2014)。
Datta,S。等。在斯坦福医学上加速临床数据科学的新范式。预印本https://arxiv.org/abs/2003.10534(2020)。
Stang,P。E。等。推进科学进行主动监视:观察医学成果伙伴关系的基本原理和设计。安.实习生。医学。 153,600 A 606(2010)。
Steyaert,S。等。癌症生物标志物发现的多模式数据融合,深入学习。纳特。马赫。英特尔。 5,351â362(2023)。
Ding,D。Y.,Li,S.,Narasimhan,B。&Tibshirani,R。多视图分析的合作学习。过程。国家科学院。科学。美国 119,E2202113119(2022)。
Guarrasia,V。等。生物医学应用多模式深学习中中间融合的系统回顾。预印本https://arxiv.org/abs/2408.02686(2024)。
Stahlschmidt,S。R.,Ulfenborg,B。&Synnergren,J。生物医学数据融合的多模式深度学习:评论。简短的。生物信息。 23,BBAB569(2022)。
Steinberg,E。等。语言模型是电子健康记录数据的有效表示学习技术。J.生物医学。通知。 113,103637(2021)。
Stelzer,I。A.等。母体代谢组,蛋白质组和免疫组的综合轨迹预测了劳动发作。科学。译。医学。 13,EABD9898(2021)。
Li,L.,Seno,M.,Yamada,H。&Kojima,I。贝丁素蛋白通过促进链链球菌素治疗的小鼠中内部前体前体细胞转化为α-细胞来改善葡萄糖代谢。是。J.生理学。内分泌。元数据。 第285章,E577âE583(2003)。
Piquer-Garcia等。Oncostatin M在肥胖症中葡萄糖稳态损害中的作用。J.克林.内分泌。元数据。 105,E337 e348(2020)。
Romero,R。等。在发作早期和晚期前启动前一项纵向研究之前,孕产妇血浆溶解的ST2浓度升高。J. Matern。胎儿新生儿医学。 31,418 A432(2018)。
Sasmaya,P。H.,Khalid,A。F.,Anggraeni,D.,Irianti,S。&Akbar,M。R.正常妊娠三个月的母体可溶性ST2水平的差异与先兆子痫。欧元。J.奥布泰特。妇科。重现。生物。X 13,100140(2022)。
谷歌学术一个
Gursoy,A。Y.等。妊娠并发症的头三个月C水平的预后价值。J.佩里纳特。医学。 44,295 299(2016)。
Bellos,I.,Fitrou,G.,Daskalakis,G.,Papantoniou,N。&Pergialiotis,V。血清Cystatin-C作为先兆子痫的预测因素:27个观察性研究的荟萃分析。怀孕高血压。 16,97 - 104(2019)。
Singh,H。&Aplin,J。D.子宫内膜根尖糖蛋白分析揭示了钙粘蛋白6,Desmoglein-2和Plexin B2在上皮完整性中的作用。摩尔。哼。重现。 21,81 - 94(2015)。
Metzenmacher,M。等。CFRNA用于疾病检测和监测的临床实用性:非小细胞肺癌概念研究证明。索拉克。癌症 13,2180 2191(2022)。
Sun,B。B。等。英国生物库的血浆蛋白质组学与遗传学和健康相关。自然 622,329 338(2023)。
Kotaskova,J。等。慢性淋巴细胞性白血病细胞中淋巴细胞激活基因3(LAG3)的高表达与未腐烂的免疫球蛋白可变重链区域(IGHV)基因和无治疗生存降低有关。J.莫尔。诊断。 12,328 334(2010)。
李,Y.等人。Behrt:用于电子健康记录的变压器。科学。代表。 10,7155(2020)。
崔,H.等人。SCGPT:使用生成AI建立一个为单细胞多媒体建立基础模型。纳特。方法 21,1470年1480(2024)。
Liu,K。等。使用电子健康记录进行急性肾脏损伤风险估算的转移学习的个性化模型的开发和验证。JAMA 网络。打开 5,E2219776(2022)。
De Francesco,D。等。数据驱动的新生儿健康和发病率的纵向表征。科学。译。医学。 15,EADC9854(2023)。
Seong,D。等。通过电子健康记录基础模型将临床记录解卷来产生怀孕的患者生物学特征。简短的。生物信息。 25,BBAE574(2024)。
萨德洛,C.等人。英国生物银行:一种开放获取资源,用于确定中老年各种复杂疾病的原因。公共科学图书馆医学。 12,E1001779(2015)。
Mikolov,T.,Chen,K.,Corrado,G。&Dean,J。对向量空间中单词表示的有效估计。预印本https://arxiv.org/abs/1301.3781(2013)。
Jehan,F。等。低收入国家和中等收入国家的早产的多组学表征。JAMA 网络。打开 3,E2029655(2020)。
唐,W.等人。乳腺癌的综合蛋白质转录组揭示了与亚型和存活相关的全球蛋白-MRNA一致性。基因组医学。 10,94(2018)。
Erhan,D。等。为什么无监督的预训练有助于深度学习?J.马赫.学习。资源。 11,625 - 660(2010)。
Li,X.,Grandvalet,Y。和Davoine,F。使用卷积神经网络转移学习的基线正规化计划。模式识别。 98,107049(2020)。
Mataraso,S。Samson920/彗星:Doi的Zenodo Repo。泽诺多https://doi.org/10.5281/Zenodo.13977341 (2024)。下载参考资料
这项工作得到了NIH No的支持。
R35GM138353(致N.A.),Burroughs Wellcome Fund No。1019816(致N.A. and D.S.),Dimes的三月(致D.S.,G.M.S.,B.G.和N.A.),Alfred E. Mann Foundation(N.A.Inv-037517(致N.A.,D.S。和G.M.S.)和Inv-076306(to N.A.),以及NSF GRFP No。DGE-2146755(致S.J.M.)。这项研究使用了斯坦福大学医学研究数据存储库Starr提供的数据或服务,这是一家临床数据仓库,其中包含来自斯坦福大学医疗保健,斯坦福儿童医院,大学医疗保健联盟和Packard Children Children的实时史诗数据S Health Alliance Clinics以及来自医院应用的其他辅助数据,例如放射线PAC。Starr平台由斯坦福医学研究技术团队开发和运营,并由斯坦福医学院研究办公室实现。This research also used the UK Biobank Resource under application no.106206.
道德声明
利益竞争
作者声明没有竞争利益。
同行评审
同行评审信息
自然机器智能thanks Zheng Xia and Paul Fogel for their contribution to the peer review of this work.
附加信息
Publisher’s note施普林格·自然对于已出版的地图和机构隶属关系中的管辖权主张保持中立。
补充资料
权利和权限
开放获取本文根据知识共享署名 4.0 国际许可证获得许可,该许可证允许以任何媒介或格式使用、共享、改编、分发和复制,只要您对原作者和来源给予适当的认可,并提供链接到知识共享许可证,并指出是否进行了更改。The images or other third party material in this article are included in the article’s Creative Commons licence, unless indicated otherwise in a credit line to the material.If material is not included in the article’s Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder.要查看该许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/。转载和许可
引用这篇文章
Mataraso, S.J., Espinosa, C.A., Seong, D.
等人。A machine learning approach to leveraging electronic health records for enhanced omics analysis.Nat Mach Intell(2025)。https://doi.org/10.1038/s42256-024-00974-9
已收到:
公认:
已发表:
DOI:https://doi.org/10.1038/s42256-024-00974-9