背景
胶质母细胞瘤(GBM)是一种高度侵略性的脑肿瘤,与患者预后差有关。
尽管有标准的疗法,但生存率仍然很低,强调了迫切需要新的治疗策略。高级成像技术,尤其是磁共振成像(MRI),对于评估GBM至关重要。各种致癌信号通路的破坏,例如受体酪氨酸激酶(RTK)-RAS-Extracellachular信号调节激酶(ERK)信号传导,3-激酶(PI3KS),肿瘤蛋白p53(TP53)(TP53)和神经源性局部局部蛋白质蛋白质(PI3KS),磷酸辛酶耐药酶3-激酶(PI3KS),以及Notch),有助于不同肿瘤类型的发展,每种肿瘤类型都表现出不同的形态和表型特征,可以在微观水平上观察到。但是,确定靶向治疗的遗传异常通常需要侵入性手术,从而促使探索非侵入性方法,例如放射基因组学。这项研究探讨了放射基因组学和机器学习(ML)在预测GBM患者中这些致癌信号通路方面的效用。
方法
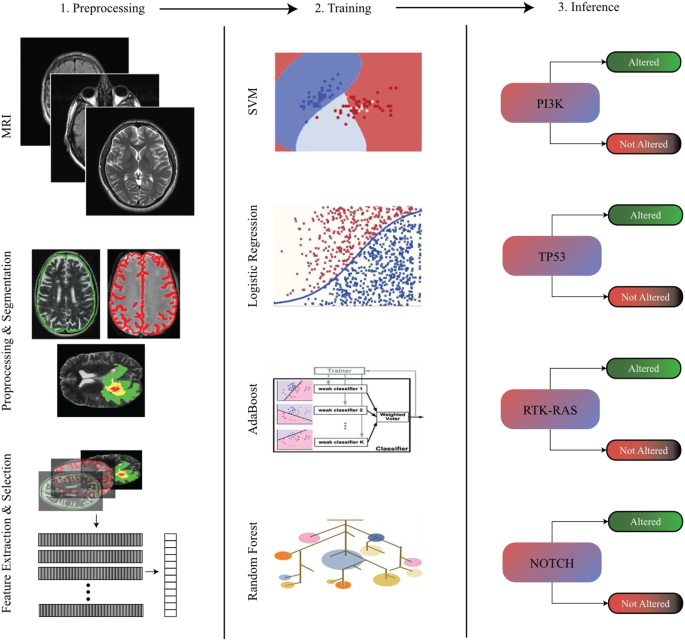
我们从BRATS-19数据集收集了术后MRI扫描(T1W,T1C,Flair,T2W),包括通过TCGA和CPTAC与遗传和临床数据相关的GBM和LGG患者的扫描。信号通路数据是从CBIOPORTAL中手动提取的。使用吡拉迪综合学从四个MRI模式中提取了放射素特征。应用了降低性降低和特征选择,并使用SMOTE解决数据不平衡。训练了五个ML模型以预测信号通路,网格搜索优化的超参数和5倍的交叉验证可确保无偏性的性能。使用测试数据的各种指标评估了每个模型的性能。
结果
我们的结果表明,大多数信号通路与从MRI扫描得出的放射线特征之间存在正相关。最佳模型获得了高AUC得分,即RTK-RAS为0.7,PI3K为0.8,TP53为0.75,Notch为0.4,因此,在准确预测了来自放射性特征的致癌信号通路中,ML模型在准确预测了个性化的肿瘤信号传导方面的潜力治疗方法和改善患者预后。
结论
我们提出了一种新的方法,用于通过将放射基因组数据与机器学习模型整合到胶质母细胞瘤(GBM)中的致癌信号通路(GBM)中的非侵蚀性预测。这项研究有助于推进GBM管理中的精确医学,强调将放射线学与基因组数据相结合以更好地了解肿瘤行为和治疗反应的重要性。
介绍
胶质母细胞瘤(GBM)是一种源自大脑内神经组织的强大恶性肿瘤,在肿瘤学上提出了重大挑战,代表了所有原发性脑肿瘤的几乎一半。可悲的是,GBM患者的预后仍然黯淡,平均存活周期为15至20个月,仅5%的人在3至5年内显示出缓解的潜力[1,,,,2,,,,3,,,,4,,,,5,,,,6]。GBM细胞表现出较高的攻击性,对标准疗法的抗性以及快速增殖,主要定位于跨区域和额叶[1,,,,3,,,,7]。GBM被世界卫生组织(WHO)归类为4年级的恶性肿瘤,由于其毁灭性影响,尤其是在年龄较大的人中,在64岁时的诊断中位数中位数[8,,,,9,,,,10,,,,11]。近年来,美国GBM的发病率一直在上升,估计有14,190例新病例在2022年报告,反映了与往年相比增加了12.1%。尽管遵循涉及手术切除,化学疗法和放疗的标准临床方案,但GBM患者仍面临仅6.9%的5年生存率的微薄,这强调了迫切需要新的治疗策略[12,,,,13]。高级成像技术(例如磁共振成像(MRI))对于准确评估胶质母细胞瘤至关重要。虽然这些成像方式大大改善了诊断,但鉴定有针对性治疗的遗传异常通常需要侵入性手术,并承担重大风险[14,,,,
15,,,,16]。针对这些挑战的一种有希望的解决方案在于放射基因组学,这是一种非侵入性方法,该方法分析了从各种成像方式中提取的放射素特征以检测和鉴定肿瘤。放射基因组学将遗传学与放射素学和人工智能(AI)结合在一起,通过鉴定遗传特征,为靶向治疗和精确医学提供了新的可能性。通过分析从医学成像获得的表型数据,放射基因组学提供了对肿瘤行为和治疗反应的见解[17,,,,18,,,,19,,,,20]。为了增强放射基因组学的预测能力,机器学习算法(AI的一个子集)由于他们能够在分析大型,复杂的数据集以识别模式和预测结果的能力方面发挥了至关重要的作用。当应用于放射基因组数据时,ML模型可以将成像特征与特定的遗传异常联系起来。最近的研究表明,肿瘤表型和基因型特征之间的直接关联,激发了从医学成像技术中提取基因型信息的兴趣[21,,,,22,,,,23]。
致癌信号通路,调节必需的细胞过程,例如生长,生存和凋亡。信号通路(例如RAS-ERK)(细胞外信号调节激酶)和PI3K(磷脂酰肌醇3-激酶)的破坏会导致异常细胞行为,并有助于癌症进展[24,,,,25,,,,26]。使用精确药物对这些途径的有针对性抑制提供了个性化的治疗策略[27,,,,28]。胶质母细胞瘤是癌症研究中强大的对手,有效地利用了各种信号通路来促进癌细胞的持续生长和生存。这个协调的网络在维持肿瘤的侵略行为方面起着至关重要的作用。主要途径之一是PI3K,它在胶质母细胞瘤的病理生理学中起着至关重要的作用。由于PTEN(磷酸酶和Tensin同源性)抑制肿瘤基因的丧失,PI3K途径通常变得过度活跃,从而促进肿瘤生长[29]。此外,在神经胶质发生中必不可少的Notch(神经源基因座Notch同源蛋白)与TP53途径相互作用,影响细胞死亡[30,,,,31,,,,32,,,,33]。不幸的是,在GBM中经常破坏TP53(肿瘤蛋白53),这是一种重要的肿瘤[29]。此外,通常在胶质母细胞瘤中改变的受体酪氨酸激酶(RTK-RAS)途径与过度活跃的RTK受体有关,进一步加剧了疾病的复杂性[34,,,,35,,,,36]。此外,Wnt信号对于中枢神经系统发育至关重要,在GBM中的调节失调,尤其是在胶质母细胞瘤干细胞(GSC)中,这有助于肿瘤的生长,并使其对药物具有抵抗力[37,,,,38,,,,39,,,,40]。传统上,确定这些途径取决于基因表达分析,这是一个费力和资源密集的过程[41]。但是,放射线学的出现在革新GBM管理方面具有巨大的希望。通过利用MRI等成像方式,放射素学促进了基本分子过程的非侵入性阐明,潜在地提供了个性化的治疗策略并大大提高了患者的预后。在这种情况下,我们的研究探讨了放射基因组学和机器学习(ML)在预测GBM中的致癌信号通路方面的实用性。通过从MRI扫描中提取放射线特征并将其与基因组数据集成在一起,我们旨在确定成像表型和遗传谱之间的关联,主要集中于与神经胶质发生有关的关键信号通路。通过先进的ML算法,我们试图开发准确识别这些途径的预测模型,从而为个性化的治疗方法提供了信息并改善患者的结果。
方法论
MRI扫描和信号通路的数据收集
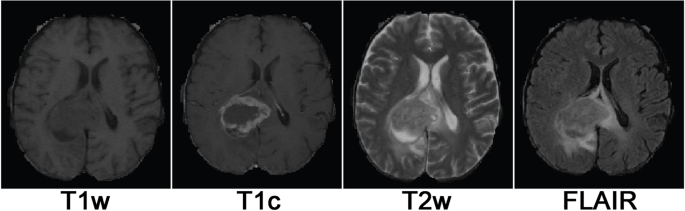
我们的实验设置是通过从各种方式中获取多个参数的术后扫描开始的,包括T1加权(T1W),对比增强的T1-加权(T1C),流体侵入的反转反恢复(FLAIR)和T2-WEIGHTEING(T2W)(T2W))图像(图 1)。这些扫描来自Brats-19数据集,该数据集由宾夕法尼亚大学的生物医学图像计算与分析中心(CBCIA)提供。42,,,,43,,,,44]。该数据集包括从诊断为GBM和低级神经胶质瘤(LGG)的患者获得的MRI扫描,并伴随着公共可访问的基因组和其他临床数据,可以通过癌症基因组图(TCGA)等平台访问[TCGA)[[TCGA)[35]和临床蛋白质组学分析联盟(CPTAC)[45]。CBCIA提供了一个文件名映射,将提供的扫描与TCGA和CPTAC门户中的患者标识符相关联,从而促进了从替代来源获得遗传和临床信息的访问。相关患者的信号通路是从CBIOPORTAL收集的。该平台提供了跨不同数据集的基因组配置文件的交互式访问,并托管了与这些配置文件相关的信号通路数据集。由于没有用于访问路径数据集的API,因此使用Web刮板手动提取路径数据。此提取是从GBM TCGA Pancancer Atlas(研究ID:GBM_TCGA_PAN_CAN_CAN_ATLAS_2018),GBM CPTAC(研究ID:GBM_CPTAC_2021)和大脑较低级glioma tcga pcaga pcancer(GBM_CPTAC_2021)和DATAS datas_pan_pan_pan_pan_pan_pan_pan_pan_pan_pan_pan_pan_pan_pan_antllan。如果在这些数据集中无法使用某些途径的情况下,则可以从TCGA FireHose Legacy数据集(研究ID:GBM_TCGA,LGG_TCGA)获得。
尽管训练套件涵盖了LGG的MRI扫描,但值得注意的是,GBM和LGG具有独特的功能和某些差异。然而,尽管有这些差异,但两种类型的神经胶质瘤也表现出重叠的成像特征并具有特定的遗传特征。两种肿瘤均显示T2加权扫描中的信号强度增加,在T1加权成像时强度降低,并在使用对比剂时表现出对比度的增强[46]。在这两种情况下,MRI扫描都通过表现出肿瘤质量以外的异常信号强度来表明肿瘤的扩散[47]。它们显示出遗传突变的相似性[48,,,,49],并且在信号通路的畸变中也有相似之处[50,,,,51]。
MRI扫描的放射素特征提取
使用为每个受试者提供的分割掩码从四个MRI模式中提取了一组放射线特征。提取过程是利用吡啶组学[52],其中包括符合成像生物标准标准化计划(IBSI)的特征定义[53]。IBSI标准化特征定义并提供了参考值,以验证放射线软件,增强可重复性并促进放射线研究的临床翻译。提取的特征集经过标准化,以在[0,1]范围内实现正态分布和归一化。但是,大量的提取特征,数量超过一千多个(> 1000)可能会导致维度的诅咒。[[54]。通过生成的功能面板上的功能工程进行了降低尺寸和最相关特征的最相关特征。
数据平衡
为了解决数据集中的数据不平衡问题,我们采用了合成少数民族过采样技术(SMOTE)[55]。Smote利用K-Nearest邻居算法在给定矢量空间中识别少数族裔类中的相邻特征向量。随后,它沿着连接少数族裔的这些相邻特征向量的线生成综合数据点。这导致了类之间扩展且狭义的决策边界,从而增强了分类模型的性能。新创建的实例保留在原始特征空间中,维护数据集的特性和分布。此外,生成的数据引入了更概括的模型的额外决策边界,这与随机过度采样等方法不同,这些方法仅复制现有实例。
选择和培训
选择了基于ML的五个监督分类模型,并使用提供的功能集来预测五个信号通路。选定的算法包括逻辑回归分类器(LRC),支持向量机(SVM),随机森林分类器(RFC),Adaboost分类器(ABC)和K-Nearest neight neighbor Classifier(KNN)。为了提高模型的预测准确性,我们采用了网格搜索超参数调整,旨在确定每种算法的最佳超参数配置。这些模型经过了5倍的交叉验证,以确保更精确和无偏的绩效评估。随后,在单独的,看不见的测试集上评估了具有最合适的高参数设置的模型。使用各种评估指标来识别每个信号通路的最佳表现算法。
结果
我们使用四种分类算法通过交叉验证来检查我们的实验结果:RFC,SVC,ABC和LRC。目的是在三个不同的数据集上检测五个特定的信号通路(Wnt,PI3K,TP53,RTK-RAS,Notch):over_sample,subly_sample和under_sample_pure。我们的方法涉及采用5倍交叉验证技术,并根据准确性,精度,召回和F1得分评估算法性能。
MRI扫描和分割标签
TCGA的GBM和LGG患者的多机构术前MRI扫描(n= 167)和cptac(nCBCIA发布的BRATS-19数据集可用= 19)。除了包含T1,T2,对比度T1和Flair 3D MRI量外,数据集还包括经验丰富的神经放射学家的分割标签。这些分割标签包括增强肿瘤部分(ET),坏死和非增强肿瘤部分(净)和周围肿瘤性水肿的注释。这些扫描已经进行了各种预处理步骤,包括颅骨划痕,共同注册和插值,以达到1毫米佣的分辨率。
收集信号通路和通过MRI扫描映射
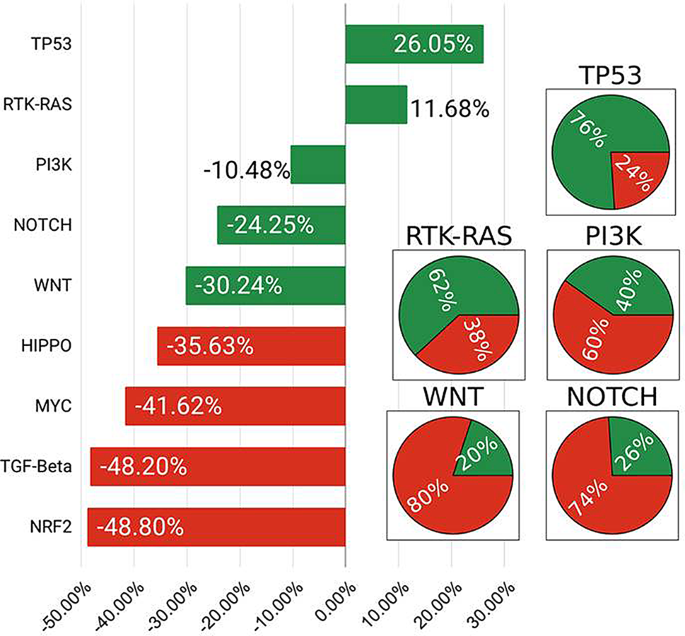
表中列出的九个致癌信号通路 1从cbioportal提取特征集。然而,两种途径NRF2和TGF-区没有显示任何改变,因此被排除在进一步分析之外。如图所示 2,途径改变的分布是不平衡的,观察到过多或稀缺的变化。在多数族裔和少数族裔之间存在显着差异的情况下,ML算法倾向于将其分类结果偏向多数级别,从而导致偏差的结果[56]。尽管达到高精度,但该算法可能无法在其他性能指标(例如灵敏度(回忆)或F1得分)方面发挥最佳性能。尽管表现出色,但Smote可能无法有效处理数据中的严重失衡。为了应对这一挑战,选择了最小不平衡(<30%)的前四个致癌信号通路进行模型训练。这些途径包括PI3K,TP53,RTK-RAS,WNT和Notch信号通路,已证明会显着影响GBM [29,,,,30,,,,31,,,,32,,,,33,,,,34,,,,36,,,,37,,,,38,,,,39,,,,40,,,,41]。
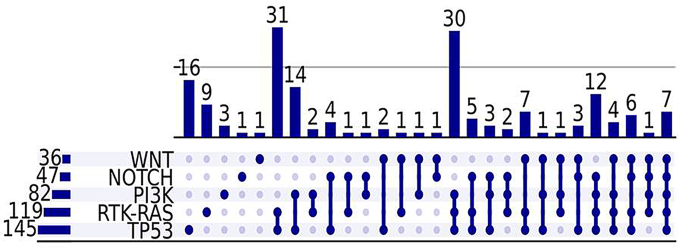
RTK-RAS,PI3K,Notch,TP53和Wnt信号通路与它们的相互作用和交叉调节相互联系,在开发和推进GBM方面起着重要作用[29,,,,30,,,,31,,,,32,,,,33,,,,34,,,,36,,,,37,,,,38,,,,39,,,,40,,,,41]。认识到这些信号通路并理解它们之间的串扰对于管理和推进目标疗法至关重要。在图2中的一个沮丧情节中描绘了跨受试者的信号通路之间的关系。 3。
放射线特征面板的推导
从遵循IBSI标准的101个标准功能得出的1284个特征的全面放射线特征集,从四个成像序列(T1,T2,T1C,Flair)及其相应的分割掩码中获得。提取的特征包括一阶,体积和基于强度的纹理特征,分为一阶,形状,GLCM,GLDM,GLRLM,GLRLM,GLSZM和NGTDM,并在表格中列出了所有107个功能 2。表2从MRI扫描中提取的特征的全面列表,用于类别形状,一阶,GLCM,GLDM,GLRLM,GLSZM和NGTDM减少维度和特征选择
随后,使用默认参数设置的Sklearn中的随机森林算法评估了每个训练集的特征重要性,以识别前10个最重要的特征。
为每种途径选择的前10个功能显示在表中 3。
创建数据集
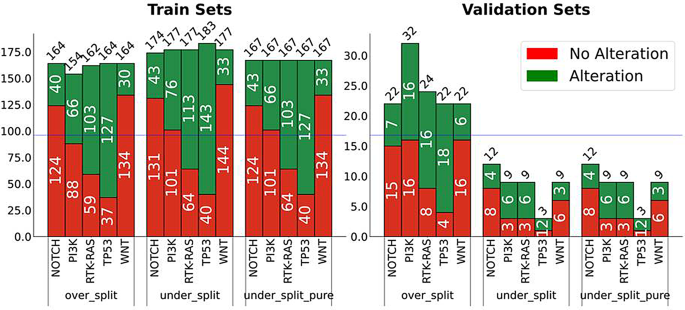
在预处理,功能工程和数据拆分之后,最终数据队列由TCGA的167名受试者(GBMâ= 98,LGGâ=â= 69)和CPTAC的19个受试者组成。将CTPAC的19位受试者放在一边以进行数据验证是适合的;但是,由于CPTAC数据集的信号传导途径的严重失衡,实验是在三个单独的数据集中进行的,这是通过将同时分配到训练和验证集中的三个单独的数据集中(图。 4)。
-
我)
在Over_Split数据集中,通过将CPTAC数据集中的样本与TCGA的一些样本组合来创建验证集。TCGA样品的选择取决于TCGA中少数类别的样品的可用性。有特定的标准用于构建此验证集。如果CPTAC数据集中的少数类别代表TCGA-GBM中的多数类,则将培训集中的所有少数族裔类样本纳入验证集中。同样,如果TCGA-GBM和CPTAC之间的少数族裔类是一致的,则选择来自TCGA-GBM的样品将其包含在验证集中。转移到验证设置的案件数量取决于TCGA-GBM中多数类与少数类别的比率。如果该比率小于0.11,则将三个案例移至验证集;否则,移动五个案件以确保平衡表示。这样做是为了平衡班级分布。
给出\(\:T = {)作为代表TCGA-GBM和CPTAC的两组\(\:\三角洲\:\)为了识别集合的类,使用以下方程生成验证集。
$ \:valset = \ left \ {\ begin {array} {c} \ begin {aligned} c \ cup \:\ left \ left \ {t:\:t \:t \:\:\ in \:{t} _ {maj} _ {maj}\:和\:\ weled | t \ right | = \ left | {c} _ {maj} \ right | - \ weft | {c} _ {min} \ right | \ right | \ right \},\ cr \ \ cr \ \:如果\:\ delta \:\ left({t} _ {maj} \右){t:\:t \:\ in \:{t} _ {min} \:and \:\:\ left | t \ t \ right | = 3 \ right \},\:如果{t} _ {maj} \ right)\ cr = \ delta \:\ left({c} _ {maj} \ right)\:and \:\ frac {\ frac {\ left | {t} _ {min} _ {min} \ right|} {\左|min} \:and \:\ weled | t \ right | = 5 \ right \},\:if \ \:\ delta \:\ left({t} _ {maj} _ {maj} \ right)\ cr = \ delta \:\ left({c} _ {maj} \ right)\:\:\ frac {\ left | {t} _ {min} \ right |} {\ left | t \ cup \ cup \:g \ right | right |}>0.11 \ end {Aligned} \ end {array} \ right。$$
-
ii)
Under_Split数据集具有一个验证集,其中包含CPTAC样本,使用不采样技术平衡。这涉及将多数级样本的数量限制为少数族裔的两倍。然后,将来自多数类的其余样本用于增加训练集的大小,从而增强训练过程。
-
iii)
Under_SPLIT_PURE数据集包含一个验证集,该验证集包含CPTAC样本,该集合通过不采样来平衡。这意味着将多数级样本的数量限制在少数群体中的两倍,并丢弃任何剩余样本。
使用K折交叉验证评估ML模型
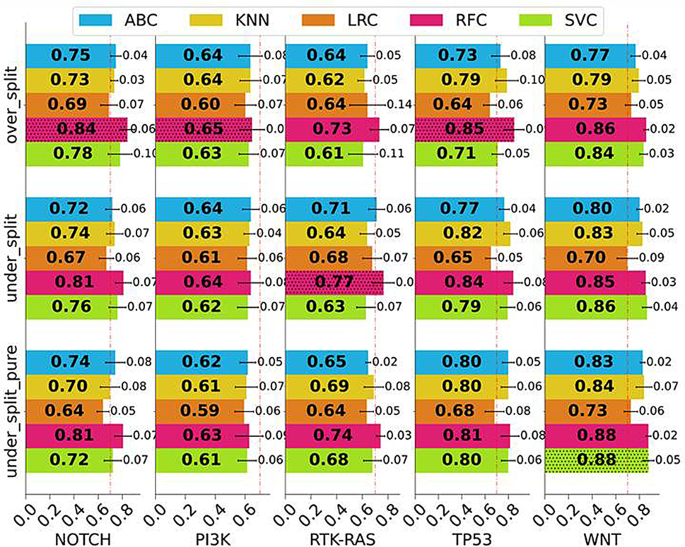
训练了五种ML算法,通过训练从GBM MRI扫描的分割标签中提取的各种放射线特征来检测五个基本的致癌信号通路。这些模型使用五倍的交叉验证进行了培训,以评估所采用方法的普遍性。在表格中列出了所有三个数据集中每个模型的5倍交叉验证的平均结果 4并在图中进一步可视化以清晰 5。除准确性外,还选择了ROC_AUC分数以对模型性能进行更全面的评估。这里的平均结果展示了使用网格搜索通过彻底的超参数调整实现的最佳参数。表中概述了负责这些结果的精确的超参数配置 5。
关于看不见数据的模型验证
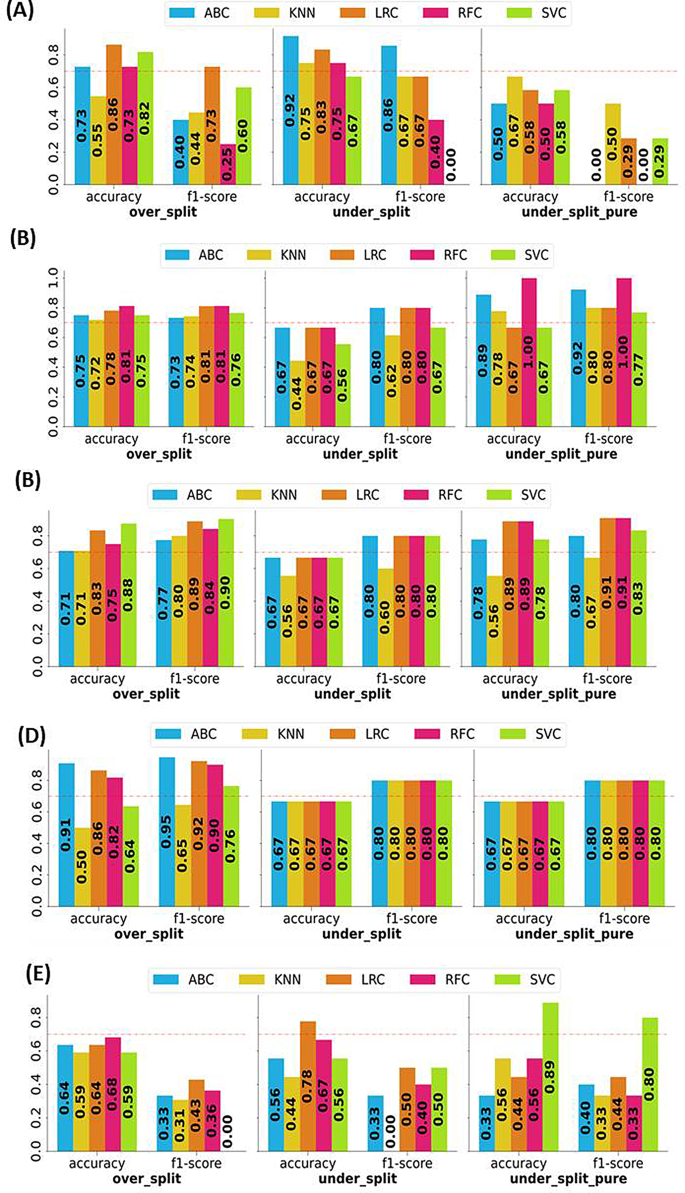
在列出的测试集上进一步验证了在每个信号通路上执行最佳的模型,以评估模型的普遍性。选择以下指标以了解一般预测能力,展示不平衡数据的性能,并观察正确的预测率和错误分类之间的权衡。在处理中度至重度数据失衡时,准确性可能不是分类问题中最合适的度量。在这种情况下,精度,回忆(特异性)和F1得分对模型的性能进行了更精确的评估。基于准确性和F1得分的算法的比较可视化图表如图所示。 6。
讨论和结论
了解RTK-RAS,PI3K,Notch和TP53等信号通路的互连性对于理解它们如何影响GBM的发展和进步至关重要。检测这些途径及其相互交流对于管理现有治疗和推进目标疗法至关重要。有趣的关联也可以在我们的数据中看到,并在图2中的不满图中显示。 3。PI3K,RTK-RAS或TP53改变在每种情况下不超过3例,而在9例中分别观察到Notch途径,在16例中,WNT途径分别观察到。相反,PI3K,RTK-RAS和TP53通常共同出现,并且观察到各种组合。例如,在31个实例中发现了RTK-RAS和TP53的独特组合,TP53在14例中只有PI3K,而在30例中,所有三种组合在一起。这三个途径在神经胶质成众所周知[35,,,,57,,,,58]。除了同时存在已建立的途径外,在35例病例中,在TP53途径的旁边发现了在p53基因中诱导p53基因凋亡的途径(如Notch)所识别的情况。这些途径之间的相互作用得到了广泛的研究和记录[33,,,,59]。值得注意的是,在总共167例案件中,所有四个途径都存在。
该研究突出了仔细选择算法,确保数据质量并在旨在旨在途径检测的无线电基因组研究中进行彻底的特征工程的重要性。每个途径都显示出不同的相互作用,并在这方面提出了具体的挑战。各种ML模型都表明评估得分有所不同,这表明特定的途径可能比其他途径更复杂的分类任务。例如,对于较低的精度,回忆和F1得分值证明,模型对模型进行准确分类似乎更困难。班级分布的变化也会影响模型性能,因为类别的类别表现出来,引入了学习中的偏见。像Notch和PI3K一样,特定的途径似乎比其他途径对模型构成了更大的挑战,这反映在较低的精度,回忆和F1得分值中。这表明这些途径内的分类边界更为复杂。
不同ML算法之间观察到的不同性能与每个途径和数据集大小所面临的不同挑战相对应。集合方法,尤其是随机森林,在不同情况下表现出一致的性能,表明它们的潜力是可靠的基线模型。TP53途径以其作为肿瘤抑制剂的功能而闻名,得出了令人兴奋的发现。当应用于OPER_SPLIT数据集时,ABC算法显示出显着的精度,精度和F1得分,表明其在检测此途径方面的功效。但是,算法的功效在under_split和uster_split_pure数据集上急剧下降,大多数算法显示出异常低的精度和回忆,这可能是由于最小的测试集构成了只有三个样品点。Conversely, cross-validation accuracy on these two datasets remained consistently above 0.70, except for LRC.
The RTK-RAS pathway, characterized by its intricate network of interactions, displayed diverse performance across different datasets. In the over_split dataset, the SVC exhibited a balanced performance. However, except KNN, all algorithms failed to identify any true negatives, classifying all nine samples as positives, leading to 100% recall but zero precision. This discrepancy contrasts with the cross-validation outcomes on the training set, suggesting that none of the models have overfit and have not achieved successful generalization.
The PI3K pathway, which plays a crucial role in cell growth and survival, exhibited relatively consistent performance trends on under_split_pure and over_split datasets. In the case of over_split, the RFC achieved consistent results across all metrics, reflecting its robustness. However, on the under_split dataset, all algorithms faced challenges detecting any true negatives primarily due to possible class imbalance issues. Interestingly, the RFC algorithm excelled on the under_split_pure dataset, with all 9 cases correctly detected. The NOTCH signaling pathway is known for its significance in gliomagenesis. Across all datasets, we observed consistently higher performance among all the algorithms over all three datasets in the prediction of NOTCH Pathway. The LRC displayed exceptional precision and recall on the over_split and under_split datasets. In contrast, all algorithms failed to perform significantly on the under_split_pure dataset.
The WNT signaling pathway, critical for cell differentiation in the central nervous system, presented diverse performance across algorithms. We had the highest class imbalance and the least number of alterations among the pathways, which performed relatively poorly on all the datasets. RFC showed variable performance across datasets, while the LRC achieved high precision and recall on the under_split dataset. The SVC demonstrated strong performance on the under_split_pure dataset, indicating its capability to handle class imbalance effectively. This research revealed insights into utilizing machine learning models with radiomic data to forecast specific oncogenic signaling pathways. The findings underscore the impact of dataset size, class distribution, and feature complexity on model effectiveness. By considering these elements, we can enhance our prediction algorithms, fostering a deeper understanding of employing AI in radiomics to elucidate the interactions among different signaling pathways and their influence on tumor phenotypic traits.
While this study highlights the potential of using artificial intelligence and radiomics to predicting oncogenic signaling pathways in glioblastoma, the study relies on publicly available datasets, such as BRATS-19 and TCGA, which may not represent the full heterogeneity of glioblastoma cases. Limited sample sizes, particularly for certain signaling pathways, have introduced class imbalance. Despite using techniques like SMOTE to address this issue, synthetic data generation may not fully capture the complexity of real-world tumor biology, potentially affecting model performance on underrepresented pathways. Furthermore, while cross-validation was used to mitigate overfitting, external validation on independent datasets could confirm the models’ reliability and applicability in diverse clinical settings.
Predicting oncogenic signaling pathways from radiomic features holds promise for advancing genomic diagnosis faster and more cost-effectively. Invasive diagnostic procedures for brain tumors, such as brain biopsies, entail additional risks, making the timely and accurate genetic profiling of specimens crucial for targeted therapeutic interventions in Glioblastoma cases. The study offers a non-invasive approach to identifying oncogenic signaling pathways, which can guide personalized therapeutic strategies which is clinically significant since this advancement could reduce reliance on invasive diagnostic procedures like biopsies, thereby mitigating associated risks. Our study deployed four machine-learning models to forecast four oncogenic signaling pathways using MRI scans from the TCGA-GBM dataset (Fig. 7)。Our findings revealed a positive correlation between the radiomic features extracted from MRI scans and oncogenic signaling pathways in GBM.With adequate data, manual feature extraction could be bypassed, leading to the development of a more generalized multi-label deep learning model capable of predicting additional signaling pathways.We intend to expand this research by developing a multi-label deep learning model that can predict a broader spectrum of signaling pathways.Future applications could also include extending the research beyond glioblastoma to other cancer types, which could help in improving patient outcomes in diverse clinical contexts.
支持数据的可用性
不适用。
参考
Grochans S, Cybulska AM, SimiÅ„ska D, Korbecki J, Kojder K, Chlubek D, Baranowska-Bosiacka I. Epidemiology of glioblastoma multiforme–literature review.癌症。2022;14(10):2412.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Lu I-N, Dobersalske C, Rauschenbach L, Teuber-Hanselmann S, Steinbach A, Ullrich V, Prasad S, Blau T, Kebir S, Siveke JT.Tumor-associated hematopoietic stem and progenitor cells positively linked to glioblastoma progression.纳特社区。2021;12(1):3895.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Kim HJ, Park JW, Lee JH.Genetic architectures and cell-of-origin in glioblastoma.前Oncol。2021;10:615400.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Melhem JM, Detsky J, Lim-Fat MJ, Perry JR.Updates in IDH-wildtype glioblastoma.神经治疗学。2022;19(6):1705–23.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Tykocki T, Eltayeb M. Ten-year survival in glioblastoma.系统评价。J Clin Neurosci。2018;54:7–13.
文章一个 PubMed一个 Google Scholar一个
Patel NP, Lyon KA, Huang JH.The effect of race on the prognosis of the glioblastoma patient: a brief review.Neurol res。2019;41(11):967–71.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Bai J, Varghese J, Jain R. Adult glioma WHO classification update, genomics, and imaging: what the radiologists need to know.顶部宏伟的岩膜成像。2020;29(2):71–82.
文章一个 PubMed一个 Google Scholar一个
Louis DN, Perry A, Wesseling P, Brat DJ, Cree IA, Figarella-Branger D, Hawkins C, Ng H, Pfister SM, Reifenberger G. The 2021 WHO classification of tumors of the central nervous system: a summary. Neurooncology. 2021;23(8):1231–51.
CAS一个 Google Scholar一个
McNeill KA. Epidemiology of brain tumors. Neurol Clin. 2016;34(4):981–98.
文章一个 PubMed一个 Google Scholar一个
Ostrom QT, Gittleman H, Farah P, Ondracek A, Chen Y, Wolinsky Y, Stroup NE, Kruchko C, Barnholtz-Sloan JS. CBTRUS statistical report: primary brain and central nervous system tumors diagnosed in the United States in 2006–2010. Neurooncology. 2013;15(suppl2):ii1–56.
Chakrabarti I, Cockburn M, Cozen W, Wang YP, Preston-Martin S. A populationâ€based description of glioblastoma multiforme in Los Angeles County, 1974–1999. Cancer: Interdisciplinary Int J Am Cancer Soc. 2005;104(12):2798–806.
文章一个 Google Scholar一个
Ostrom QT, Gittleman H, Truitt G, Boscia A, Kruchko C, Barnholtz-Sloan JS. CBTRUS statistical report: primary brain and other central nervous system tumors diagnosed in the United States in 2011–2015.Neuro-oncology2018, 20(suppl_4):iv1-iv86.
Ostrom QT, Price M, Neff C, Cioffi G, Waite KA, Kruchko C, Barnholtz-Sloan JS.CBTRUS Statistical Report: primary brain and other Central Nervous System tumors diagnosed in the United States in 2015–2019.Neuro oncol。2022;24(Suppl 5):v1–95.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Bi WL, Hosny A, Schabath MB, Giger ML, Birkbak NJ, Mehrtash A, Allison T, Arnaout O, Abbosh C, Dunn IF. Artificial intelligence in cancer imaging: clinical challenges and applications. Cancer J Clin. 2019;69(2):127–57.
文章一个 Google Scholar一个
Hussain T, Nguyen QT. Molecular imaging for cancer diagnosis and surgery. Adv Drug Deliv Rev. 2014;66:90–100.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Chen C-C, Hsu P-W, Wu T-WE, Lee S-T, Chang C-N, Wei K-c, Chuang C-C, Wu C-T, Lui T-N, Hsu Y-H.Stereotactic brain biopsy: single center retrospective analysis of complications.Clin Neurol Neurosurg。2009;111(10):835–9.
文章一个 PubMed一个 Google Scholar一个
Shui L, Ren H, Yang X, Li J, Chen Z, Yi C, Zhu H, Shui P. The era of radiogenomics in precision medicine: an emerging approach to support diagnosis, treatment decisions, and prognostication in oncology.前Oncol。2021;10:570465.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Bodalal Z, Trebeschi S, Nguyen-Kim TDL, Schats W, Beets-Tan R. Radiogenomics: bridging imaging and genomics. Abdom Radiol. 2019;44(6):1960–84.
文章一个 Google Scholar一个
Sundaram M, McGuire MH, Herbold DR.Magnetic resonance imaging of soft tissue masses: an evaluation of fifty-three histologically proven tumors.麦克谐调成像。1988;6(3):237–48.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Upadhyay N, Waldman A. Conventional MRI evaluation of gliomas.Br J radiol。2011;84(specialissue2):S107–11.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Zinn PO, Singh SK, Kotrotsou A, Hassan I, Thomas G, Luedi MM, Elakkad A, Elshafeey N, Idris T, Mosley J. A coclinical radiogenomic validation study: conserved magnetic resonance radiomic appearance of periostin-expressing glioblastoma in patients and xenograft型号。Clin Cancer Res。2018;24(24):6288–99.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Pei L, Vidyaratne L, Rahman MM, Iftekharuddin KM. Context aware deep learning for brain tumor segmentation, subtype classification, and survival prediction using radiology images. Sci Rep. 2020;10(1):19726.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Li K, Xiao J, Yang J, Li M, Xiong X, Nian Y, Qiao L, Wang H, Eresen A, Zhang Z. Association of radiomic imaging features and gene expression profile as prognostic factors in pancreatic ductal adenocarcinoma. Am J Translational Res. 2019;11(7):4491.
CAS一个 Google Scholar一个
Sever R, Brugge JS. Signal transduction in cancer. Cold Spring Harbor Perspect Med. 2015;5(4):a006098.
文章一个 Google Scholar一个
Villanueva A, GarcÃa C, Paules AB, Vicente M, MegÃas M, Reyes G, de Villalonga P, Agell N, LluÃs F, Bachs O. Disruption of the antiproliferative TGF-β signaling pathways in human pancreatic cancer cells.癌基因。1998;17(15):1969–78.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Mendoza MC, Er EE, Blenis J. The Ras-ERK and PI3K-mTOR pathways: cross-talk and compensation.趋势生物化学科学。2011;36(6):320–8.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Samimi H, Fallah P, Sohi AN, Tavakoli R, Naderi M, Soleimani M, Larijani B, Haghpanah V. Precision medicine approach to anaplastic thyroid cancer: advances in targeted drug therapy based on specific signaling pathways. Acta Medica Iranica 2017:200–8.
Zhu P, Aliabadi HM, UludaÄŸ H, Han J. Identification of potential drug targets in cancer signaling pathways using stochastic logical models. Sci Rep. 2016;6(1):23078.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Mischel PS, Nelson SF, Cloughesy TF.Molecular analysis of glioblastoma: pathway profiling and its implications for patient therapy.癌症生物。2003;2(3):242–7.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Pearson JR, Regad T. Targeting cellular pathways in glioblastoma multiforme. Signal Transduct Target Therapy. 2017;2(1):1–11.
文章一个 Google Scholar一个
Bazzoni R, Bentivegna A. Role of notch signaling pathway in glioblastoma pathogenesis.癌症。2019;11(3):292.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Gaiano N, Fishell G. The role of notch in promoting glial and neural stem cell fates.Annu Rev Neurosci。2002;25(1):471–90.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Yang X, Klein R, Tian X, Cheng H-T, Kopan R, Shen J. Notch activation induces apoptosis in neural progenitor cells through a p53-dependent pathway.开发生物。2004;269(1):81–94.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Sanchez-Vega F, Mina M, Armenia J, Chatila WK, Luna A, La KC, Dimitriadoy S, Liu DL, Kantheti HS, Saghafinia S. Oncogenic signaling pathways in the cancer genome atlas.细胞。2018;173(2):321–37.e310.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
1 CGARNTDUMSMRFABD, 5 EUVMEGBDJMMGOJJ, 8 HFHMTLN, 11 MACCAKAYWBO. 13 UoCSFVSBMPM: Comprehensive genomic characterization defines human glioblastoma genes and core pathways.自然2008, 455(7216):1061–1068.
Fonseca COd, Linden R, Futuro D, Gattass CR, Quirico-Santos T. Ras pathway activation in gliomas: a strategic target for intranasal administration of perillyl alcohol. Arch Immunol Ther Exp. 2008;56:267–76.
文章一个 Google Scholar一个
Lee Y, Lee J-K, Ahn SH, Lee J, Nam D-H.WNT signaling in glioblastoma and therapeutic opportunities.实验室投资。2016;96(2):137–50.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Komiya Y, Habas R. Wnt signal transduction pathways.器官发生。2008;4(2):68–75.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Guan R, Zhang X, Guo M. Glioblastoma stem cells and wnt signaling pathway: molecular mechanisms and therapeutic targets. Chin Neurosurgical J. 2020;6:1–6.
文章一个 Google Scholar一个
Zuccarini M, Giuliani P, Ziberi S, Carluccio M, Di Iorio P, Caciagli F, Ciccarelli R. The role of wnt signal in glioblastoma development and progression: a possible new pharmacological target for the therapy of this tumor.基因。2018;9(2):105.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Kabir MH, Patrick R, Ho JW, O’Connor MD. Identification of active signaling pathways by integrating gene expression and protein interaction data. BMC Syst Biol. 2018;12:77–87.
文章一个 Google Scholar一个
Menze BH, Jakab A, Bauer S, Kalpathy-Cramer J, Farahani K, Kirby J, Burren Y, Porz N, Slotboom J, Wiest R. The multimodal brain tumor image segmentation benchmark (BRATS). IEEE Trans Med Imaging. 2014;34(10):1993–2024.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Bakas S, Akbari H, Sotiras A, Bilello M, Rozycki M, Kirby JS, Freymann JB, Farahani K, Davatzikos C. Advancing the cancer genome atlas glioma MRI collections with expert segmentation labels and radiomic features. Sci data. 2017;4(1):1–13.
文章一个 Google Scholar一个
Bakas S, Reyes M, Jakab A, Bauer S, Rempfler M, Crimi A, Shinohara RT, Berger C, Ha SM, Rozycki M. Identifying the best machine learning algorithms for brain tumor segmentation, progression assessment, and overall survival prediction in the BRATS challenge. arXiv Preprint arXiv:181102629 2018.
Whiteaker JR, Halusa GN, Hoofnagle AN, Sharma V, MacLean B, Yan P, Wrobel JA, Kennedy J, Mani D, Zimmerman LJ.CPTAC Assay Portal: a repository of targeted proteomic assays.NAT方法。2014;11(7):703–4.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Haydar N, Alyousef K, Alanan U, Issa R, Baddour F, Al-Shehabi Z, Al-Janabi MH. Role of Magnetic Resonance Imaging (MRI) in grading gliomas comparable with pathology: a cross-sectional study from Syria. Annals Med Surg. 2022;82:104679.
文章一个 Google Scholar一个
Guillevin R, Herpe G, Verdier M, Guillevin C. Low-grade gliomas: the challenges of imaging. Diagn Interv Imaging. 2014;95(10):957–63.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Aoki K, Nakamura H, Suzuki H, Matsuo K, Kataoka K, Shimamura T, Motomura K, Ohka F, Shiina S, Yamamoto T. Prognostic relevance of genetic alterations in diffuse lower-grade gliomas. Neurooncology. 2018;20(1):66–77.
CAS一个 Google Scholar一个
Paugh BS, Qu C, Jones C, Liu Z, Adamowicz-Brice M, Zhang J, Bax DA, Coyle B, Barrow J, Hargrave D. Integrated molecular genetic profiling of pediatric high-grade gliomas reveals key differences with the adult disease.J Clin Oncol。2010;28(18):3061.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Nasrolahi A, Azizidoost S, Radoszkiewicz K, Najafi S, Ghaedrahmati F, Anbiyaee O, Khoshnam SE, Farzaneh M, Uddin S. Signaling pathways governing glioma cancer stem cells behavior.细胞信号。2023;101:110493.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Nakada M, Kita D, Watanabe T, Hayashi Y, Teng L, Pyko IV, Hamada J-I.Aberrant signaling pathways in glioma.癌症。2011;3(3):3242–78.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Van Griethuysen JJ, Fedorov A, Parmar C, Hosny A, Aucoin N, Narayan V, Beets-Tan RG, Fillion-Robin J-C, Pieper S, Aerts HJ.Computational radiomics system to decode the radiographic phenotype.癌症。2017;77(21):e104–7.
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Zwanenburg A, Leger S, Vallières M, Löck S. Image biomarker standardisation initiative. arXiv Preprint arXiv:161207003 2016.
Limkin EJ, Sun R, Dercle L, Zacharaki EI, Robert C, Reuzé S, Schernberg A, Paragios N, Deutsch E, Ferté C. Promises and challenges for the implementation of computational medical imaging (radiomics) in oncology.安·恩科尔(Ann Oncol)。2017;28(6):1191–206.
文章一个 CAS一个 PubMed一个 Google Scholar一个
Chawla NV, Bowyer KW, Hall LO, Kegelmeyer WP. SMOTE: synthetic minority over-sampling technique. J Artif Intell Res. 2002;16:321–57.
文章一个 Google Scholar一个
Krawczyk B. Learning from imbalanced data: open challenges and future directions. Progress Artif Intell. 2016;5(4):221–32.
文章一个 Google Scholar一个
Venkatesan S, Lamfers ML, Dirven CM, Leenstra S. Genetic biomarkers of drug response for small-molecule therapeutics targeting the RTK/Ras/PI3K, p53 or rb pathway in glioblastoma. CNS Oncol. 2016;5(2):77–90.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Han S, Wang P-F, Cai H-Q, Wan J-H, Li S-W, Lin Z-H, Yu C-J, Yan C-X.Alterations in the RTK/Ras/PI3K/AKT pathway serve as potential biomarkers for immunotherapy outcome of diffuse gliomas.老化。2021;13(11):15444.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Dotto GP.Crosstalk of notch with p53 and p63 in cancer growth control.Nat Rev Cancer。2009;9(8):587–95.
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
资金
This study was funded, in part, by a Research Grant (Grant number: ID No. 2022–16465) from the Indian Council of Medical Research (ICMR) Govt. of India, New Delhi to Muzafar A. Macha. Promotion of University Research and Scientific Excellence (PURSE) (SR/PURSE/2022/121) grant from the Department of Science and Technology, Govt. of India, New Delhi to the Islamic University of Science and Technology (IUST), Awantipora.
道德声明
道德批准并同意参加
不适用。
同意出版
不适用。
竞争利益
作者宣称他们没有竞争利益。
附加信息
Publisher’s note
关于已发表的地图和机构隶属关系中的管辖权主张,Springer自然仍然是中立的。
电子补充材料
以下是电子补充材料的链接。
权利和权限
开放访问This article is licensed under a Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License, which permits any non-commercial use, sharing, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons licence, and indicate if you modified the licensed material.您没有根据本许可证的许可来共享本文或部分内容的改编材料。The images or other third party material in this article are included in the article’s Creative Commons licence, unless indicated otherwise in a credit line to the material.If material is not included in the article’s Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder.要查看此许可证的副本,请访问http://creativecommons.org/licenses/by-nc-nd/4.0/。重印和权限
引用本文
Ahanger, A.B., Aalam, S.W., Masoodi, T.A.
等。Radiogenomics and machine learning predict oncogenic signaling pathways in glioblastoma.J Transl Med23 , 121 (2025). https://doi.org/10.1186/s12967-025-06101-5下载引用
:2024年11月26日
:2025年1月8日
:2025年1月27日
:https://doi.org/10.1186/s12967-025-06101-5关键字