迅速工程对生物医学文献中蛋白质 - 蛋白质相互作用识别的大语言模型的影响
作者:Lin, Yi-Hsuan
介绍
蛋白质相互作用(PPI)在各种生理过程中至关重要,例如基因表达,信号转导和凋亡,这直接影响健康和疾病1。由突变或感染等因素引起的PPI的畸变与癌症等疾病密切相关2,,,,3。此外,PPI会影响各种行业,如它们在食品加工中的作用,在食品加工中可以看出,辣椒蛋白等酶对分解小麦面筋蛋白至关重要4,在蛋白质相互作用影响果实成熟的农业中5。PubMed的广泛数据库举例说明了生物医学文献的激增,对研究人员提出了有效挖掘和提取可操作的见解的挑战。为了帮助解决这个机会,自然语言处理的进步(NLP),例如命名实体识别(NER),关系提取(RE)6和问答(QA)7,具有巨大的潜力。引入基于变压器的模型,例如Bert8在2018年彻底改变了这一领域,并导致了Biobert等专业版本9,Scibert10和临床伯特11,增强特定于任务的模型性能。
从文献中提取蛋白质 - 蛋白质相互作用(PPI)的早期方法包括基于模式的,共同出现和机器学习策略。基于模式的技术涉及手动构建规则以识别蛋白质对,并提供了一种基础方法12,,,,13,而同时出现的方法利用单句内蛋白质对的发生来促进提取,尽管其能力限制了捕获复杂关系14。诸如机器学习之类的高级方法使用了结合语言和结构元素以增强PPI提取的全面功能集,尽管这些方法通常会面临与数据中与结构相似性相关的挑战15,,,,16。此外,基于内核的技术,例如子树17,子集树18,部分树19,光谱树20,富含特征的树21内核有效地使用高维句子特征来优化提取过程22,,,,23;复合内核方法通过整合多种内核类型来完善这种方法,以增强文本信息的提取和分析24,,,,25,,,,26。此外,梯度树的发展模型LPGBOOST的开发标志着计算效率通过浅数据表示的重大进步27。此外,神经网络架构,尤其是卷积(CNN)和经常性神经网络(RNN),通过自主提取特征提取来发挥关键作用;这增加了内核方法并提高了PPI提取任务的总体功效28,,,,29,,,,30。基于变压器的BERT模型的集成代表了PPI提取方法的进一步演变。这些模型将深度学习技术与词汇和句法处理结合在一起,以显着推进该领域,并更加精确,有效地提取蛋白质相互作用31,,,,32。但是,尽管域特异性模型具有有效性,但它们的更广泛的应用和有效的实施通常受到基础计算机科学知识的要求的限制。相比之下,大型语言模型(LLM)的开发(例如2022年推出的OpenAi的Chatgpt)提供了一种有希望的选择。这些模型,在大量数据集中进行了预训练,能够在无需特定于任务的微调的情况下在各个领域中产生上下文相关的响应33。此外,Openai的生成预训练的变压器系列中的LLM已从GPT-1演变为更健壮和多模式的GPT-4,这在理解和生成语言方面表现出广泛的功能34,,,,35。这些高级模型可以无缝支持一系列应用,而无需特定域的调整36,,,,37。
与开源语言模型相比,需要在培训和预测的计算方法方面的技术专业知识,Chatgpt和Gemini等专有LLM38使非技术用户能够通过精心设计的提示有效地获得特定于域的参考结果。这种能力可以显着加速药物开发等领域的研究过程39。在这项研究中,我们使用蛋白质 - 蛋白质相互作用(PPI)预测任务作为案例研究,以评估专有LLMS在响应专门设计的提示方面的性能,以模拟生物医学研究人员可用的援助程度。我们的目标不仅是在专门任务中评估这些模型的内在功能和局限性,而且还探讨了迅速优化可以提高其性能的程度。总而言之,该分析旨在对专有LLM在处理复杂的生物学数据中的适用性提供更清晰的观点,并在此过程中提供对生命科学中AI技术实际部署的宝贵见解。
方法
蛋白质蛋白质相互作用(PPI)任务定义
蛋白质 - 蛋白质相互作用(PPI)的定义可能会根据不同应用所需的实体的概念描述而有所不同。自1970年代成立以来40DNA,RNA和蛋白质的相互依赖性已得到充分认识。但是,生物医学文献中通常采用的以基因为中心的表示倾向于加剧这些分子实体之间的歧义。为了提高生物相互作用建模的准确性,许多研究扩大了蛋白质实体的定义,包括基因和RNA41,,,,42,,,,43,,,,44。因此,PPI数据集根据其预期应用在对蛋白质实体的定义上有所不同。此外,蛋白质之间相互作用的定义涵盖了多种观点,从直接物理接触到文本来源中描述的更广泛的上下文关联。为了减轻由顺序实体识别和关系预测引起的错误传播,大多数PPI数据集提供了预通量的实体,这有助于随后的建模工作。在这项研究中,我们遵守每个数据集提供的实体注释并进行句子级预测。在及时开发过程中,该模型呈现了一对包含这些实体的命名实体和句子,并且它的任务是预测蛋白质对是否相互作用。然后将包含相互作用实体对的句子归类为积极实例,而没有相互作用的句子则被归类为负面实例。如果句子包含多个实体对,则将分别评估每个对关系。例如,如果确定SIGK和GERE没有相互作用,则该实例被标记为负相互作用,而如果将SIGK和YKVP识别为在同一句子中的相互作用,则将其标记为正。补充表中提供的示例中说明了这一点1。
评估数据集和实验设置
我们使用六个PPI基准数据集来评估我们的提示策略:LLL,IEPA,HPRD50,瞄准,生物学和PEDD。LLL数据集源自Logic 2005(LLL05)挑战中的学习语言,并来自Medline数据库,重点是仅使用77个句子提取蛋白质/基因相互作用,即有限的信息。45。IEPA数据集包含486个从PubMed摘要中提取的句子46。HPRD50数据集包括来自人类蛋白参考数据库(HPRD)的50个随机摘要,总共有145个句子,由Priginer预先标记的蛋白质/基因和人类专家注释的相互作用关系13。目标数据集包含来自PubMed的200个摘要,并用实体注释及其相互作用关系47。生物学数据集包含1100个句子,并使用相互作用蛋白(DIP)的数据库收集,以识别与交互实体相关的PubMed搜索输入;选定的句子包含多对互动实体48。我们还使用了最近发布的PEDD数据集,该数据集源自AICUP 2019年的竞赛,重点介绍2015年以后发布的PubMed的摘要以及影响因素大于5的期刊,以确保更高质量和最新信息49。这些数据集的正面和负PPI实例的原始分布在补充表的原始数据列中介绍2。值得注意的是,由于单个句子可能包含正面和负面实例,因此总句子计数大大低于正面和负面实例的总和。
我们的实验工作流程遵循两阶段评估方法。在第一阶段,我们从五个基准数据集(LLL,IEPA,HPRD50,Aimed和Bioinfer)中选择了代表性样本进行使用GPT-3.5和GPT-4的及时设计和初步评估。在第二阶段,我们使用所有六个数据集(包括PEDD)进行了全面的绩效评估,包括GPT-3.5,GPT-4和Gemini 1.5(flash和Pro版本)。所有实验均使用相应的LLM API在Python中实现。为了确保一致的评估,我们要求所有LLM以标准化的JSON格式输出其预测,这有助于对我们使用的三个标准指标进行系统的计算:召回,精度和F1-分数。精确度量通过计算所有被确定为阳性的实例中的真实案例的比例来衡量积极预测的准确性;这表明模型的能力避免了误报。回忆量化了系统正确识别的真实阳性的比例,从而反映了模型捕获所有相关相互作用的能力。f1-Score通过将精度和回忆组合为单个值来平衡模型的整体性能。为了调查提供其他上下文是否可以改善基于LLM的PPI提取,我们尝试了多句输入(从1到5个句子)。理论上添加上下文信息可能会增强模型的推断相互作用的能力,但它也引入了额外噪声的可能性,可能会影响预测可靠性。我们的结果表明,多句子输入导致不同模型和数据集的性能波动,这表明较长的输入并不能始终如一地提高预测精度。为了确保方法学的一致性和可重复性,我们的主要评估是使用单句输入进行的。结果部分提供了对该比较的详细分析。
方法论框架概述
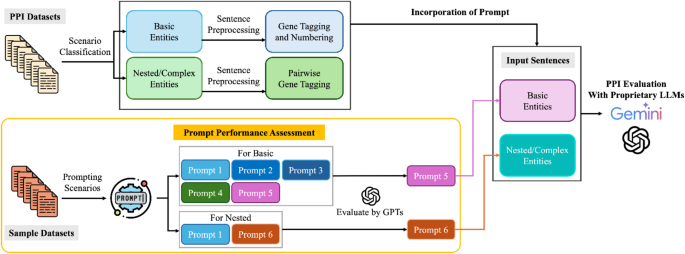
这项研究提出了一种系统的方法,可以通过精心设计的提示策略来评估专有LLMS在PPI检测中的能力。我们的方法包括三个主要组成部分:(1)提示工程和优化,(2)上下文感知提示选择,以及(3)跨多个数据集的系统评估。在迅速的工程阶段,我们开发了六个不同的促使方案,从基本互动查询到复杂的实体标签格式逐渐增加了复杂性。这些场景旨在评估不同级别的输入结构和上下文信息如何影响模型性能。提示在两个关键维度上有所不同:实体标记程度(从未标记到用数值标识符进行全面标记)和查询语句的特异性(从简单的交互查询到结构化的JSON输出请求)。上下文感知的选择机制可以基于句子特征来实现自适应及时部署。该组成部分专门解决了生物医学文献中各种蛋白质实体表示所带来的挑战,包括复杂的实体(例如“ ARP2/3复合物”)。选择过程确保最合适的提示应用于每个特定上下文。为了进行系统评估,我们实现了一个综合的工作流,该工作流程顺序处理PPI数据集,并结合了成对蛋白质标记和上下文特定的提示选择。这种结构化方法允许在不同数据集进行一致的评估,同时适应其独特特征。以下各节详细介绍了这些组件,首先是对提示方案的深入检查,然后是高级工作流,将这些提示集成到连贯的PPI检测系统中。
提示场景
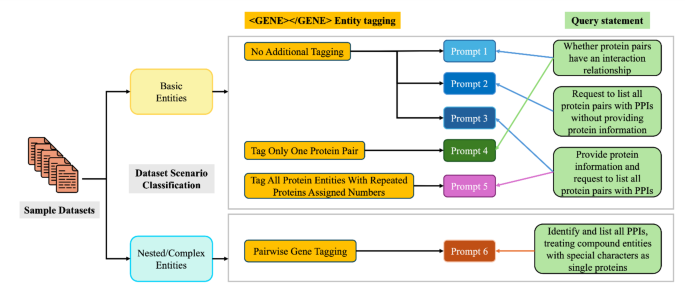
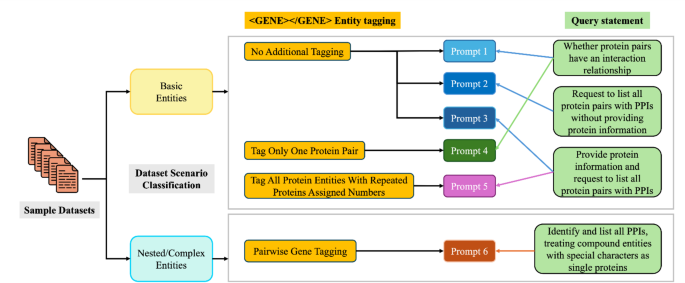
为了系统地评估PPI提取,我们设计了一种全面的提示策略,如图所示。 1。该策略将输入数据分为两种主要类型:基本实体和嵌套/复杂实体。对于基本实体,我们制定了三个不同的实体标记策略:(1)直接处理原始文本的其他标签;(2)仅标记一个蛋白质对,重点是特定的蛋白质对;(3)标记所有具有重复蛋白质数字的蛋白质实体,该蛋白质具有全面的蛋白质标记。这些标记策略与逐渐复杂的查询陈述相结合:从基本的相互作用验证(提示1)到识别没有明确蛋白质信息的PPI(提示2-3),再到完整的蛋白质吸引的PPI识别(提示4-5)。对于嵌套/复杂实体,我们实现了一种专门的成对基因标记方法(提示6),以处理具有特殊字符的复合实体。这些复杂情况的更详细的处理方法将在稍后引入。所有标记的实体都使用“ <gene> </gene>”标记模式来标准化跨不同提示场景的蛋白质实体的识别。
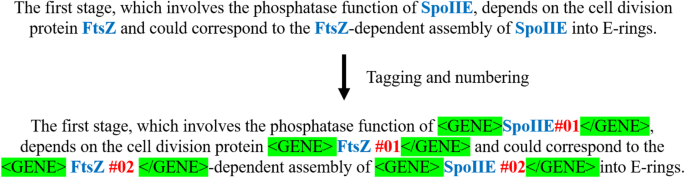
补充人物1展示提示1和3的输入输出对的示例,其中用户是指定的输入和助理GPT型号的输出。在句子'lll.d2.s2 - 存在三个蛋白质实体中,这导致了三对通过排列和组合产生的输入问题。示例(a)在补充数字中1说明了提示1的单个提示对,而示例(b)演示了提示3在同一句子中的应用。鉴于提示2和3引起了模型的PPI结果的多个输出,因此已增强了及时描述,以包括以JSON格式的响应请求,以简化后续的结果汇总和分析。图中的输入侧的描述显示了提示3和提示2之间的差异。通过列举句子中的所有蛋白质实体名称,目的是评估该模型是否可以增强其关联判断能力。在与提示3相同的查询框架内,最复杂的提示提示5需要明确,全面的实体标记和输入句子编号。图 2说明输入句子标记的示意图。图2
选定的句子符合至少生成3至6个正/负实例的标准,以维持样品集中的均匀分布。样品数据集的统计数据如图2所示。将示例数据集应用于GPT-3.5和GPT-4模型以选择最可靠的提示。
在数据检查期间,我们建立了一种系统的方法,以解决超越基本预定义提示的复杂实体方案。我们的分析揭示了需要专业处理的三种主要模式:基于符号的复合实体,关键字指示的复合物和嵌套实体。首先,我们实施了基于符号的过滤以识别复合蛋白质实体。包含具有“/”或“”符号的蛋白质名称的句子被标记为复合蛋白质实体。例如,“ ARP2/3复合物”代表两个不同的蛋白质,Arp2â和“ Arp3”,起作用为统一复合物。这些标记的蛋白质名称进行了其他分析,以确保模型的准确解释。其次,我们为复杂命名实体开发了基于关键字的识别。包含特定关键字,例如“复杂”,“亚基”或“组件”的句子被确定为潜在的复合命名实体。例如,“ ARP2/3复合物结合肌动蛋白丝”例证了这种情况。这些句子通过LLM收到了专门的处理,以防止因缩写或命名变化引起的预测错误。第三,我们解决了嵌套实体方案,其中短形式和描述性的长格式蛋白质名称出现在同一上下文中。例如,在...两个跨膜受体,p75神经营养蛋白受体和p140TRK(TRKA)酪氨酸激酶受体,...“,p75及其更长的形式” p75神经营养蛋白受体出现为嵌套实体。为避免手动干预,我们采用了一个基因缩写识别模块来自动检测和处理这些嵌套和复合术语。及时的GPT指示GPT指示GPT在蛋白质名称中,而不是仅仅考虑完整的AbbReviations,而不是PPI识别。
对数据集的统计分析表明,这些复杂的实体模式仅在目标和生物促进数据集中出现,占每个数据集中大约23%的实例。为了解决这些方案,我们开发了提示6,它通过纳入处理复合条款,嵌套实体及其变化的全面准则来建立在提示1的基础上。实体标记策略需要列出所有蛋白质实体名称并系统地检查蛋白质对,句子处理仅在评估所有可能的蛋白质对组合后才考虑完成。图 3使用生物学数据集示例演示了这种方法,带下划线的文本突出了复合实体描述,并比较了不同LLM模型的预测。图3
描述PPI的句子通常由于研究主题,作者写作风格和实验类型的差异而差异很大,这使得它们挑战了分类。
为了应对这些挑战,我们开发了一个综合框架,如图所示。 4,首先是使用示例数据集的及时设计和评估。该框架从提示场景(图中的左下方)启动,图中显示了详细的设计原理。 1)。在此阶段,输入句子是根据实体复杂性分类的:基本实体和复杂实体(包括复杂,化合物或嵌套结构)。对于每个类别,候选人提示使用GPT模型进行评估,以确定针对该特定情况的最有效提示。结果是基本实体提示5,并提示复杂实体的6。在及时评估后,所有PPI数据集句子都会根据其实体复杂性进行场景分类。然后,使用适当的标记策略来处理这些句子,以突出基因位置:基本实体经历基因标记和编号,而复杂的实体是通过成对基因标记处理的。最后,这些处理后的句子与它们相应的方案特定提示结合使用,以使用包括GPT和Gemini模型在内的专有LLMS进行性能评估。这种系统的方法可确保适当处理各种PPI描述,同时根据实体复杂性和句子结构优化及时选择。可以通过这种结构化方法对预测的性能进行全面评估。
结果
确定候选提示
实验的主要目的是确定最佳的一般提示(提示1 -5),以帮助模型辨别不同的PPI。为了实现这一目标,我们采用了补充图中划定的PPI样本数据集2作为评估的焦点并对GPT-3.5和GPT-4模型进行了一般提示的绩效评估。这些发现,如表所示 1,一贯证明,提示5在两个模型迭代中在样本数据集中表现出卓越的性能。根据补充数字3,提示5的设计策略可帮助GPT准确地识别PPI预测期间具有相同名称的多个蛋白质,并随后根据内容关联列出实际交互组。随后利用提示5所证明的功效,我们研究了同时输入多个句子对GPT模型性能的影响(表 2)。研究结果表明,不仅证明了GPT-4的优越性能,而且还表明了单句输入方法对模型性能的影响更加一致。
为了评估提示6的性能,这是专门设计用于处理涉及嵌套蛋白质实体的病例的,我们创建了一个评估样本集。该集合由针对目标和生物学数据集的10个句子组成。在瞄准数据集中,有15个积极实例和27个负面实例,而生物学数据集包含42个正实例和30个负面实例。该样本组成可以平衡评估及时识别相关实体的有效性。鉴于其类似的查询语句结构,提示1作为评估的基线。表中列出的结果 3表明在GPT-3.5和GPT-4型号中,提示6的提示均优胜1。这突出了提供有关增强大语言模型性能的复合术语信息的有益影响。
PPI数据集的性能评估
基于先前建立的及时过滤过程,我们使用最终提示5和6评估了六个PPI数据集的GPT-3.5,GPT-4和Google Gemini 1.5(闪存和Pro版本)。 4在所有模型中呈现比较结果。值得注意的是,Gemini 1.5 Pro在大多数数据集中表现出卓越的性能,达到了最高的F1 - LLL(90.3%),IEPA(68.2%),HPRD50(67.5%)和PEDD(70.2%)数据集中的分数。GPT-4在LLL数据集中表现出竞争性表现(87.3%F1 - 分数),同时通常在其他数据集中保持稳定的性能。与其他模型相比,GPT-3.5表现出一致但性能相对较低。跨数据集的模型性能的变化反映了其特征的固有挑战。尽管最小的LLL数据集在所有型号中的性能最高(范围为79.2%至90.3%F1 - 分数),这表明其尺寸较小,并且可能更直接的实体关系有助于更好的模型理解。相比之下,诸如目标,生物学和PEDD之类的更复杂的数据集在所有模型中显示出较低的性能,1 - 原始数据的评分通常在37.9至70.2%之间。这种性能模式突出了这些数据集中复杂的实体功能和丰富内容所带来的挑战。
此外,我们在所有模型中都确定了预测积极输入实例的偏见,从而导致歧视能力降低,而在处理仅处理负面实例时。为了解决此限制,我们排除了在绩效评估过程中仅包含数据集中的负面实例的句子。表的精制数据列 4在此修改后演示模型性能。LLL数据集缺乏这样的实例,维护了其原始分布,并被排除在精制评估之外。在这种情况下,所有模型均表现得提高了精度和F1 - 精制数据集的分数。例如,在IEPA数据集中,Gemini 1.5 Pro实现了F1 - 分别为89.7%,而GPT-4和GPT-3.5分别达到86.4%和84.7%。跨模型和数据集的这种一致的改进进一步验证了我们对模型积极实例偏见的观察结果,并证明了我们的改进策略的有效性。
讨论
LLM在生物关系提取上的应用既带来了有希望的机会,又带来了显着的挑战。我们对PPI预测中LLM的全面评估揭示了有关模型性能,局限性和实际影响生物医学研究界的几个关键见解。在讨论中,我们首先系统地检查模型性能和数据集特性之间的关系,然后对模型特定结果进行详细分析。然后,我们在探索未来的方向和潜在应用之前应对关键局限性和挑战。我们的分析表明,不同模型和数据集的有效性不同,具有特别值得注意的性能模式。最引人注目的观察结果是在所有模型中始终在LLL数据集上达到的卓越性能,而F1 - 分数从79.2%到90.3%,双子座1.5 Pro达到最高F1 - 分数为90.3%。这种出色的性能可以归因于几个因素,包括较小的尺寸,简单的注释方案和更简单的语法结构,这为模型理解提供了一个较不具有挑战性的环境50。这些特定于数据集的特征构成了理解整个评估套件中模型性能的更广泛模式的基础。
跨数据集的性能变化揭示了对模型行为和局限性的关键见解。除了定量绩效指标之外,我们的语言分析还发现了语义解析和参考理解方面的重大挑战。我们观察到,当与复杂的相互作用相关的语言结构面对时,LLMS表现出明显的错误分类敏感性。两个说明性的例子强调了这些解释性挑战。首先,考虑从Bioinfer.D245摘录的句子”现在,我们在这里确定了α-catenin中的相互互补结合位点,该结合位点介导了其与<基因>β-catenin</基因>和<基因>plakoglobin</基因>。“词汇提示,例如”调解“ 和 ”相互作用“引起了模型驱动的误解,错误地表明β-catenin和plakoglobin之间存在直接的PPI关系。批判性地,模型忽略了Alpha-Catenin与这些实体分别相互作用的细微现实。它的相互作用,“代表了当代LLM的重大解释性脆弱性。摘自Aimed.d182的第二个例子进一步强调了LLMS中参考歧义的挑战。具体而言,该句子是该句子MDM2癌蛋白是一种细胞抑制剂<基因>p53</基因>肿瘤抑制剂可以结合<基因>p53</基因>并下调其激活转录的能力演示重复基因提及如何需要准确的核心分辨率来保留语义清晰度。在这种情况下,两个p53实体显然缺乏蛋白质 - 蛋白质相互作用。该代词明确地提到了句子的主要主题,”MDM2癌蛋白". Nevertheless, the models consistently failed to discern these critical contextual nuances, erroneously predicting an interconnection. The marked contrast between simpler datasets like LLL and more complex ones such as AIMed and BioInfer highlights how sentence complexity and grammatical structure significantly impact extraction accuracy. This is particularly evident in AIMed’s sophisticated sentence constructions and nested entities, which resulted in notably lower precision scores of 26.6% and 23.8% for GPT-3.5 and GPT-4, respectively. The representation of biological entities across different datasets also proves to be a crucial factor, with BioInfer’s complex compound entities and abbreviations significantly challenging model comprehension, as reflected in its lower F1-scores ranging from 45.3 to 55.3% in the raw data. In contrast, datasets featuring more straightforward entity mentions, such as HPRD50, demonstrated more robust performance with F1-scores between 63.6 and 67.5%.Additionally, our analysis uncovered a consistent bias across all models toward predicting positive instances, leading us to refine our evaluation approach by excluding sentences containing solely negative instances.This refinement resulted in substantial performance improvements, as evidenced by IEPA’s F1-scores increasing from 65.6 to 84.7% for GPT-3.5 and from 64.0 to 86.4% for GPT-4;this underscores the significant impact of data distribution on model behavior.
Gemini 1.5 Pro demonstrated superior performance across most datasets, which can be attributed to its advanced model architecture and optimization.This superior performance was particularly evident for the LLL (90.3% F1-score), IEPA (89.7% F1-score in refined data), and HPRD50 (87.1% F1-score in refined data) datasets.GPT-4 showed competitive performance, particularly in the LLL dataset (87.3% F1-score), while maintaining stable performance across other datasets.GPT-3.5, while showing consistent performance, generally achieved lower scores compared to other models.To contextualize these results within the current research landscape, a recent study by Rehana et al.51evaluated GPT and BERT models on PPI tasks, achieving F1-scores of 86.49% (GPT-4 on LLL), 71.54% (GPT-4 on IEPA), and 65.0% (GPT-4 on HPRD50) using strategies such as protein dictionary normalization and protein masking.While their approach relies on sophisticated preprocessing techniques, our prompt design approach achieves comparable or superior results while focusing solely on sentence structure, and in so doing, offers a more user-friendly approach for non-technical biomedical researchers.Further analysis of the LLMs’ behavior in specific linguistic contexts revealed additional performance patterns.We examined PPI sentences from the PEDD dataset containing inference-related keywords including whether, may, might, could, potential, and other similar terms;we identified 2,570 cases with 282 positive and 2,288 negative instances for evaluation.In these inferential contexts, Gemini 1.5 Pro maintained its superior performance with an F1-score of 0.2, followed by Gemini 1.5 Flash at 0.179 and GPT-3.5 at 0.163, while GPT-4 unexpectedly showed the lowest performance at 0.154.The significantly lower F1-scores in inferential contexts compared to general PPI detection suggest that LLMs struggle to interpret protein interactions when presented with hypothetical or uncertain relationships.Notably, all models exhibited high false positive rates in inferential contexts, with even the best-performing Gemini 1.5 Pro producing 1,550 false positive cases.This observation provides additional evidence of a broader pattern in LLMs’ prediction behavior that warrants further investigation, as we discuss in our analysis of the study’s limitations.
While these results demonstrate promising capabilities of LLMs in biomedical relationship extraction, several important limitations and challenges emerged from our analysis.The observed bias of LLMs towards positive PPI predictions warrants careful consideration.A comprehensive review discusses how biases can manifest in various natural language processing tasks, including literature mining, highlighting the tendency of LLMs to generate outputs that may favor certain narratives or perspectives52。The ability to accurately identify both the presence and absence of protein interactions is crucial for understanding biological systems and developing therapeutic interventions.Our findings suggest that current LLMs, despite their sophisticated architecture, may exhibit a systematic bias that could lead to false positive predictions in PPI detection tasks.This limitation could be particularly problematic in exploratory research where identifying non-interacting protein pairs is as valuable as detecting interactions.Our analysis also revealed interesting variations in how different LLMs identify potential PPIs within established complexes.For example, when analyzing the sentence "Arp2/3 complex from Acanthamoeba binds profilin and cross-links actin filaments" and subsequently asked whether Arp2 and Arp3 components interact with each other, we observed divergent interpretations: GPT-3.5 identified a positive PPI between Arp2 and Arp3 subunits within the complex, while GPT-4, Gemini 1.5 Flash, and Gemini 1.5 Pro classified it as a negative interaction. This discrepancy highlights a fundamental challenge in how LLMs process relationships between proteins that function as part of established complexes. The Arp2/3 example demonstrates that even when addressing interactions between components of well-characterized molecular assemblies, LLMs can produce inconsistent results. This variation represents an important consideration when employing LLMs for biomedical knowledge extraction, particularly for questions about the internal structure of protein complexes that may not be explicitly stated in the text.
Traditional transformer-based models (BioBERT, PubMedBERT, SciBERT) have demonstrated superior performance across these datasets, with BioBERT achieving F1-scores ranging from 74.95% (HPRD50) to 86.84% (LLL)51。Similarly, previous machine learning approaches have shown competitive results, with sdpCNN achieving 66% and 75.2% on AImed and BioInfer respectively53, and the tree-based DSTK model achieving scores ranging from 71.01% (AImed) to 89.20% (LLL)23。Based on these findings and observed limitations, we recommend that practitioners implementing LLMs for PPI detection should: (1) implement additional validation steps for negative predictions, (2) consider incorporating confidence scores or uncertainty measures in their predictions, and (3) potentially employ ensemble approaches that combine LLM predictions with traditional machine learning methods that have shown robust performance in identifying negative instances.Looking toward future developments, while the system testing results obtained through prompt design may not surpass various state-of-the-art deep learning models, LLMs offer unique advantages.Their ability to adapt prompt suitability continuously without retraining for specific task demands54and potential to outperform state-of-the-art models when fine-tuned on specific datasets55makes them particularly valuable for non-computer science experts56。Recent research has shown promising strategies for improving LLM performance in biomedical applications.Few-shot learning approaches have demonstrated comparable results to SOTA models in tasks such as NER, relation extraction, summarization, and QA55。In the Nephrology domain, chain-of-thought (CoT) strategies have successfully guided models in clinical decision support57。Future development of LLMs for biomedical applications should explicitly address the positive prediction bias, perhaps through specialized pre-training strategies or architectural modifications that better balance the model’s ability to identify both positive and negative protein interactions.LLMs have already begun to appear in highly specialized clinical research, such as extracting diagnosis information from cancer examination reports58,,,,59。Their performance on the recent PEDD dataset (achieving 70.2% F1-score compared to BioBERT’s 77.06%)49demonstrates their ability to handle contemporary biomedical literature effectively, though precise task completion still relies heavily on appropriate prompt guidance60。In an era emphasizing multi-objective applications, the integration of large language models can provide a robust foundation for development and research across various domains.Our findings suggest that when combined with effective prompt engineering strategies and domain-specific considerations, LLMs have the potential to revolutionize biomedical relationship extraction, particularly for researchers without extensive computational expertise.
结论
Verifying interactions in biomedical experiments requires precise conditions and results in complex biomedical literature and PPI datasets.This study shows that general-purpose LLMs like the GPT and Gemini models can reliably predict protein interactions and serve as valuable tools for non-experts.Using progressive prompt designs tailored to PPI dataset specificities, we addressed model biases, such as misclassifying negative instances as positive, by refining data preprocessing and prompt design to significantly enhance performance.Although the GPT and Gemini models currently lag behind traditional state-of-the-art methods, the rapid advancement of LLM technology offers a promising future.Innovative techniques, such as chain-of-thought prompts and ensemble predictions with multiple LLMs, are expected to further improve performance.The continued evolution of LLMs holds significant potential for advancing research across an ever diversifying range of fields.
数据可用性
The five benchmark PPI datasets (AImed, Bioinfer, HPRD50, IEPA and LLL) analyzed in this study are publicly available on the GitHub repository athttps://github.com/metalrt/ppi-dataset/tree/master/csv_output。Additionally, the latest PPI dataset referenced in this study, PPED, can be accessed via the AI CUP 2019 platform athttps://www.aicup.tw/ai-cup-2019。参考
Berggård, T., Linse, S. & James, P. Methods for the detection and analysis of protein–protein interactions.
蛋白质组学7 (16), 2833–2842 (2007).文章
一个 PubMed一个 Google Scholar一个 Garner, A. L. & Janda, K. D. Protein-protein interactions and cancer: targeting the central dogma.Curr。
顶部。医学化学 11(3), 258–280 (2011).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Hoffmann, M. et al.SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor.细胞 181(2), 271–280.e8 (2020).
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Agyare, K. K., Addo, K. & Xiong, Y. L. Emulsifying and foaming properties of transglutaminase-treated wheat gluten hydrolysate as influenced by pH, temperature and salt.Food Hydrocolloids 23(1), 72–81 (2009).
文章一个 CAS一个 Google Scholar一个
Wang, S. et al.Phosphorylation of MdERF17 by MdMPK4 promotes apple fruit peel degreening during light/dark transitions.植物细胞 34(5), 1980–2000 (2022).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Kim, J.-D., et al.Overview of BioNLP shared task 2011 InProceedings of the BioNLP Shared Task 2011 Workshop(Association for Computational Linguistics, 2011).
Tsatsaronis, G. et al.An overview of the BIOASQ large-scale biomedical semantic indexing and question answering competition.BMC Bioinform. 16, 1–28 (2015).
文章一个 Google Scholar一个
Devlin, J., et al.BERT:深层双向变压器的预训练,以了解语言理解。(North American Chapter of the Association for Computational Linguistics, 2019).
Lee, J. et al.BioBERT: a pre-trained biomedical language representation model for biomedical text mining.生物信息学 36(4), 1234–1240 (2020).
文章一个 MathSciNet一个 CAS一个 PubMed一个 Google Scholar一个
Beltagy, I., Lo, K. & Cohan, A. SciBERT: A pretrained language model for scientific text.在Conference on Empirical Methods in Natural Language Processing(2019)。
Alsentzer, E. et al.Publicly Available Clinical BERT Embeddings(Association for Computational Linguistics, 2019).
Huang, M. et al.Discovering patterns to extract protein–protein interactions from full texts.生物信息学 20(18), 3604–3612 (2004).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Fundel, K., Küffner, R. & Zimmer, R. RelEx—Relation extraction using dependency parse trees.生物信息学 23(3), 365–371 (2007).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Bunescu, R., et al.Integrating co-occurrence statistics with information extraction for robust retrieval of protein interactions from Medline.在Proceedings of the hlt-naacl bionlp Workshop on Linking Natural Language and Biology(2006)。
Van Landeghem, S., et al.Extracting protein-protein interactions from text using rich feature vectors and feature selection.在3rd International symposium on Semantic Mining in Biomedicine (SMBM 2008)。(Turku Centre for Computer Sciences (TUCS), 2008).
Liu, B., et al.Dependency-driven feature-based learning for extracting protein-protein interactions from biomedical text.在Coling 2010: Posters(2010)。
Vishwanathan, S. & Smola, A. J. Fast kernels for string and tree matching.ADV。神经。inf。过程。系统。 15, 569–576 (2003).
Collins, M., Parsing with a single neuron: Convolution kernels for natural language problems.Technical Report, (University of California at Santa Cruz, 2001).
Moschitti, A. Making tree kernels practical for natural language learning.在11th conference of the European Chapter of the Association for Computational Linguistics(2006)。
Kuboyama, T. et al.A spectrum tree kernel.inf。Media Technol. 2(1), 292–299 (2007).
Sun, L. & Han, X. A feature-enriched tree kernel for relation extraction.在Proceedings of the 52nd Annual Meeting of the Association for Computational Linguistics (Volume 2: Short Papers)(2014)。
Li, L. et al.An approach to improve kernel-based protein–protein interaction extraction by learning from large-scale network data.方法 83, 44–50 (2015).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Murugesan, G., Abdulkadhar, S. & Natarajan, J. Distributed smoothed tree kernel for protein–protein interaction extraction from the biomedical literature.PLoS ONE 12(11), e0187379 (2017).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Miwa, M. et al.Protein–protein interaction extraction by leveraging multiple kernels and parsers.int。J. Med。通知。 78(12), e39–e46 (2009).
文章一个 PubMed一个 Google Scholar一个
Li, L. et al.Integrating semantic information into multiple kernels for protein-protein interaction extraction from biomedical literatures.PLoS ONE 9(3), e91898 (2014).
文章一个 广告一个 PubMed一个 PubMed Central一个 Google Scholar一个
Chang, Y.-C.等。PIPE: A protein–protein interaction passage extraction module for BioCreative challenge.数据库 2016, baw101 (2016).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Warikoo, N., Chang, Y.-C.& Ma, S.-P.Gradient boosting over linguistic-pattern-structured trees for learning protein–protein interaction in the biomedical literature.应用。科学。 12(20), 10199 (2022).
文章一个 CAS一个 Google Scholar一个
Hsieh, Y.-L., et al.Identifying protein-protein interactions in biomedical literature using recurrent neural networks with long short-term memory.在Proceedings of the eighth international joint conference on natural language processing (volume 2: short papers)(2017)。
Peng, Y. & Lu, Z.Deep Learning for Extracting Protein–Protein Interactions from Biomedical literature(Association for Computational Linguistics, 2017).
书一个 Google Scholar一个
Yadav, S., et al.Feature assisted bi-directional LSTM model for protein–protein interaction identification from biomedical texts (2018).
Su, P., Peng, Y. & Vijay-Shanker, K. Improving BERT model using contrastive learning for biomedical relation extraction.在Workshop on Biomedical Natural Language Processing(2021)。
Warikoo, N., Chang, Y.-C.& Hsu, W.-L.LBERT: Lexically aware transformer-based bidirectional encoder representation model for learning universal bio-entity relations.生物信息学 37(3), 404–412 (2021).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Wu, T. et al.A brief overview of ChatGPT: The history, status quo and potential future development.IEEE/CAA J. AUTOM。罪。 10(5), 1122–1136 (2023).
文章一个 Google Scholar一个
Radford, A., et al.通过生成的预训练来改善语言理解。OpenAI preprint (2018).
Radford, A. et al.语言模型是无监督的多任务学习者。OpenAI blog 1(8), 9 (2019).
Brown, T. et al.语言模型是很少的学习者。ADV。神经。inf。过程。系统。 33, 1877–1901 (2020).
Gallifant, J. et al.Peer review of GPT-4 technical report and systems card.PLOS数字健康 3(1), e0000417 (2024).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Team, G., et al.Gemini: A family of highly capable multimodal models.ARXIV预印本arXiv:2312.11805(2023)。
Tripathi, S. et al.Large language models reshaping molecular biology and drug development.化学生物。Drug Des. 103(6), e14568 (2024).
文章一个 CAS一个 PubMed一个 Google Scholar一个
Crick, F. Central dogma of molecular biology.自然 227(5258), 561–563 (1970).
文章一个 广告一个 CAS一个 PubMed一个 Google Scholar一个
Jaeger, S. et al.Integrating protein-protein interactions and text mining for protein function prediction.BMC Bioinform. 9, S2 (2008).
文章一个 Google Scholar一个
Krallinger, M., Valencia, A. & Hirschman, L. Linking genes to literature: text mining, information extraction, and retrieval applications for biology.基因组生物。 9, 1–14 (2008).
文章一个 Google Scholar一个
Taha, K. & Yoo, P. D. Predicting the functions of a protein from its ability to associate with other molecules.BMC Bioinform. 17, 1–28 (2016).
Al-Aamri, A. et al.Constructing genetic networks using biomedical literature and rare event classification.科学。代表。 7(1), 15784 (2017).
文章一个 广告一个 PubMed一个 PubMed Central一个 Google Scholar一个
Nédellec, C. Learning language in logic-genic interaction extraction challenge.在Proceedings of the 4th Learning Language in Logic Workshop (LLL05)。Citeseer (2005).
Ding, J., et al.Mining MEDLINE: abstracts, sentences, or phrases?, InBiocomputing 2002, 326–337.(World Scientific, 2001).
Bunescu, R. et al.Comparative experiments on learning information extractors for proteins and their interactions.艺术品。Intell。医学 33(2), 139–155 (2005).
文章一个 PubMed一个 Google Scholar一个
Pyysalo, S. et al.BioInfer: a corpus for information extraction in the biomedical domain.BMC Bioinform. 8, 1–24 (2007).
文章一个 Google Scholar一个
Huang, M.-S.等。Surveying biomedical relation extraction: a critical examination of current datasets and the proposal of a new resource.简短的。Bioinform. 25(3), bbae132 (2024).
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Gajendran, S., Manjula, D. & Sugumaran, V. Character level and word level embedding with bidirectional LSTM–Dynamic recurrent neural network for biomedical named entity recognition from literature.J. Biomed。通知。 112, 103609 (2020).
文章一个 PubMed一个 Google Scholar一个
Rehana, H. et al.Evaluating GPT and BERT models for protein–protein interaction identification in biomedical text.Bioinform.ADV。 4(1), vbae133 (2024).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Guo, Y., et al.Bias in large language models: Origin, evaluation, and mitigation.ARXIV预印本arXiv:2411.10915(2024)。
Hua, L. & Quan, C. A shortest dependency path based convolutional neural network for protein-protein relation extraction.生物。res。int。 2016(1), 8479587 (2016).
Wang, J. et al.Review of large vision models and visual prompt engineering.元放射学 1, 100047 (2023).
文章一个 Google Scholar一个
Jahan, I. et al.A comprehensive evaluation of large language models on benchmark biomedical text processing tasks.计算。生物。医学 171, 108189 (2024).
文章一个 PubMed一个 Google Scholar一个
Wang, J., et al.Prompt engineering for healthcare: Methodologies and applications.ARXIV预印本arXiv:2304.14670(2023)。
Miao, J. et al.Chain of thought utilization in large language models and application in nephrology.Medicina 60(1), 148 (2024).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Choi, H. S. et al.Developing prompts from large language model for extracting clinical information from pathology and ultrasound reports in breast cancer.Radiat.Oncol。J. 41(3), 209 (2023).
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Huang, J. et al.A critical assessment of using ChatGPT for extracting structured data from clinical notes.npj Digital Med. 7(1), 106 (2024).
文章一个 Google Scholar一个
White, J., et al.A prompt pattern catalog to enhance prompt engineering with Chatgpt.ARXIV预印本arXiv:2302.11382。(2023)。
资金
This research was supported by the National Science and Technology Council of Taiwan, grant number NSTC 113-2221-E-038-019-MY3Â and NSTC 113-2627-M-A49-002, as well as from the National Health Research Institutes, under grant number NHRI-12A1-PHCO-1823244.This work was also financially supported by the Higher Education Sprout Project, funded by the Ministry of Education (MOE) in Taiwan (grant number DP2-TMU-114-A-04).
道德声明
竞争利益
作者没有宣称没有竞争利益。
附加信息
Publisher’s note
关于已发表的地图和机构隶属关系中的管辖权主张,Springer自然仍然是中立的。
电子补充材料
Below is the link to the electronic supplementary material.
权利和权限
开放访问This article is licensed under a Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License, which permits any non-commercial use, sharing, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons licence, and indicate if you modified the licensed material.您没有根据本许可证的许可来共享本文或部分内容的改编材料。The images or other third party material in this article are included in the article’s Creative Commons licence, unless indicated otherwise in a credit line to the material.If material is not included in the article’s Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder.要查看此许可证的副本,请访问http://creativecommons.org/licenses/by-nc-nd/4.0/。重印和权限
引用本文
Chang, YC., Huang, MS., Huang, YH.
等。The influence of prompt engineering on large language models for protein–protein interaction identification in biomedical literature.Sci代表15 , 15493 (2025).https://doi.org/10.1038/s41598-025-99290-4
已收到:
公认:
出版:
doi:https://doi.org/10.1038/s41598-025-99290-4