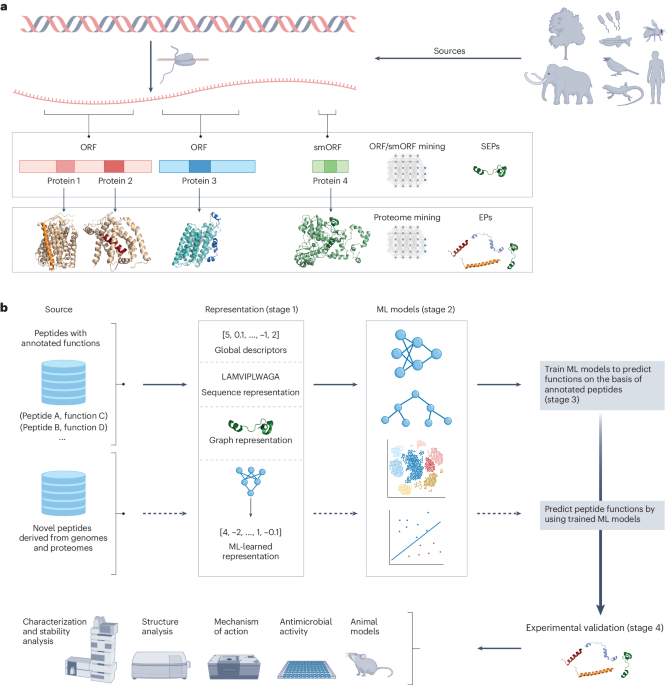
教程:使用机器学习方法来挖掘基因组和蛋白质组的指南抗生素发现
作者:de la Fuente-Nunez, Cesar
Magana,M。等。
抗菌肽在抗性时代的值。柳叶刀感染。dis。 20,E216 e230(2020)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Murray,C。J。L.等。2019年细菌抗菌耐药性的全球负担:系统分析。柳叶刀 399,629 - 655(2022)。
文章一个 CAS一个 Google Scholar一个
De la Fuente-Nunez,C.,Torres,M。D.,Mojica,F。J.&Lu,T。K.下一代精度抗菌剂:朝着个性化的传染病治疗。Curr。意见。微生物。 37,95 102(2017)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Porto,W。F.等。在番石榴抗菌肽的硅硅中,可以为肽设计提供组合探索。纳特。社区。 9,1490(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Wong,F。,de la Fuente-Nunez,C。&Collins,J。J.在与传染病的斗争中利用人工智能。科学 381,164â170(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Maasch,J。R. M. A.,Torres,M。D. T.,Melo,M。C. R.&de la Fuente-Nunez,C。通过机器学习启用的古代抗菌肽的分子去渗透。细胞宿主微生物 31,1260年1274(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Torres,M。D. T.等。在人蛋白质组中开采加密的肽抗生素。纳特。生物。工程。 6,67 - 75(2022)。
文章一个 PubMed一个 Google Scholar一个
Wan,F.,Torres,M。D. T.,Peng,J。&de la Fuente-Nunez,C。通过分子去灭绝发现深度学习的抗生素。纳特。生物。工程。 8,854 A 871(2024)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Diâ©Guez-Santana,K。&Gonzâlez-Dãaz,H。迈向机器学习发现双重抗菌药物纳米颗粒系统的发现。纳米级 13,17854年17870(2021)。
文章一个 PubMed一个 Google Scholar一个
Nocedo-Mena,D。等。通过机器学习和化学结构信息与微生物代谢网络的化学结构信息进行建模。J. Chem。inf。模型。 59,1109年1120(2019)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Liu,G。等。深度学习引导的抗生素靶向发现baumannii acinetobacter。纳特。化学生物。 19,1342 - 1350(2023)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Wong,F。等。发现具有可解释的深度学习的结构性抗生素类别。自然 626,177年185(2024)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Hughes,J。,Rees,S.,Kalindjian,S。&Philpott,K。早期药物发现原则。br。J. Pharmacol。 162,1239年1249年(2011年)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
MA,Y.等。使用深度学习从人肠道微生物组中鉴定抗菌肽。纳特。生物技术。 40,921â931(2022)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Torres,M。D. T.等。挖掘人类微生物组揭示了未开发的肽抗生素来源。细胞 187,5453 - 5467(2024)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Santos-JãºNior,C。D。等。通过机器学习,在全球微生物组中发现抗菌肽。细胞 187,3761 3778.E16(2024)。
文章一个 PubMed一个 Google Scholar一个
Pane,K。等。通过计算实验平台实现的新型隐性多功能抗菌肽的新型隐性多功能抗菌肽的鉴定。ACS合成器。生物。 7,2105 2115(2018)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Cesaro,A。等。从人血浆中加入蛋白质中的序列的合成抗生素。ACS纳米 16,1880年1895年(2022年)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Sberro,H。等。人类微生物组的大规模分析揭示了成千上万的小型新基因。细胞 178,1245年1259.e14(2019)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Li,H。等。FSPP:用于全基因组预测SMORF编码肽及其功能的工具。正面。基因。 9,96(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
托雷斯(M. D. T.J. Mol。生物。 431,3547 A3567(2019)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Torres,M。D. T.,Cesaro,A。&de la Fuente-Nunez,C。非免疫蛋白的肽通过抗菌和免疫调节特性靶向感染。趋势生物技术。 43,184年205(2025)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Yuanyuan,J。&Xinqiang,Y。从人基因组中鉴定出的菌根。J. Proteome Res。 21,865 - 873(2022)。
文章一个 PubMed一个 Google Scholar一个
Martinez,T。F.等。分析小鼠棕色和白色脂肪细胞,以鉴定代谢相关的小型ORF和功能性微蛋白。细胞代谢。 35,166 - 183.e11(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Ruiz-Orera,J。&Albã,M。M.小型开放阅读框架的翻译:调节和进化创新中的作用。趋势基因。 35,186â(2019)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Sandmann,C.-L。等。人类,年轻的微蛋白和小型肽的进化起源和相互作用从简短的开放式阅读帧翻译而成。摩尔。细胞 83,994年1011.E18(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Makarewich,C。A.&Olson,E。N.挖掘小肽。趋势细胞生物。 27,685 - 696(2017)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Vitorino,R.,Guedes,S.,Amado,F.,Santos,M。&Akimitsu,N。微肽在生物学中的作用。细胞。摩尔。生命科学。 78,3285 3298(2021)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Sousa,M。E.&Farkas,M。H.菌根。PLOS基因。 14,E1007764(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Eraslan,G.,Avsec,Ž。,Gagneur,J。&Theis,F。J.深度学习:基因组学的新计算建模技术。纳特。基因牧师。 20,389 403(2019)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
托雷斯(M. D.ACS纳米 15,2143 2164(2021)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Rombel,I。T.,Sykes,K。F.,Rayner,S。&Johnston,S。A. Orf-Finder:高通量基因识别的向量。基因 282,33â41(2002)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Bepler,T。&Berger,B。学习蛋白质语言:进化,结构和功能。细胞系统。 12,654 - 669.e3(2021)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Rives,A。等。生物结构和功能从缩放无监督的学习到2.5亿个蛋白质序列。Proc。纳特。学院。科学。美国 118,E2016239118(2021)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Madani,A。等。大型语言模型会在不同家族中产生功能蛋白序列。纳特。生物技术。 41,1099年1106(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Alley,E。C.,Khimulya,G.,Biswas,S.,Alquraishi,M。&Church,G。M.统一的统一理性蛋白质工程,基于序列的深度表示学习。纳特。方法 16,1315年1322(2019)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
De la Fuente-Nunez,C。AI在传染病中:数据集的作用。抗药性。更新。 73,101067(2024)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Pane,K。等。阳离子抗菌肽的抗菌效力可以从其氨基酸组成中预测:应用于检测神秘的抗菌肽。J.理论。生物。 419,254 265(2017)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Uniprot联盟。UNIPROT:蛋白质知识的全球枢纽。核酸res。 47,D506 d515(2019)。
文章一个 Google Scholar一个
Goldberg,K。等。蛋白酶体衍生的防御肽通过细胞自主的先天免疫。自然 639,1032â1041(2025)。
Xia,X.,Torres,M。D. T.&de la Fuente-Nunez,C。通过深度学习发现的蛋白酶体衍生的抗菌肽。预印本Biorxiv https://doi.org/10.1101/2025.03.17.643752(2025)。
Bhadra,P.,Yan,J.,Li,J.,Fong,S。&Siu,S。W. I. Ampep:使用氨基酸特性和随机森林的分布模式对基于序列的抗菌肽的预测。科学。代表。 8,1697(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Pirtskhalava,M。等。DBAASP V3:抗菌/细胞毒性活性和肽结构的数据库,作为开发新疗法的资源。核酸res。 49,D288 d297(2021)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
托伦斯(A.纳特。生物技术。 42,1179年1180(2024)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Rawlings,N。D.,Barrett,A。J.&Bateman,A。Merops:肽酶数据库。核酸res。 38,D227 d233(2010)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
De Oliveira,D。M. P.等。埃斯卡普病原体中的抗菌素抗性。临床微生物。修订版 33,E00181-19(2020)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Kawashima,S。Aaindex:氨基酸指数数据库。核酸res。 28,374 374(2000)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Wang,G.,Li,X。&Wang,Z。APD3:抗菌肽数据库作为研究和教育的工具。核酸res。 44,D1087 d1093(2016)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Kang,X。等。流水2.0,这是抗菌肽的更新数据存储库。科学。数据 6,148(2019)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Zhao,X.,Wu,H.,Lu,H.,Li,G。&Huang,Q。Lamp:链接抗微生物肽的数据库。PLOS一个 8,E66557(2013)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Jhong,J.-H。等。DBAMP 2.0:具有增强基因组和蛋白质组学扫描方法的抗菌肽的更新资源。核酸res。 50,D460 d470(2022)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
麦金尼斯(L.J.开源软件。 3,861(2018)。
文章一个 Google Scholar一个
Andaur Navarro,C。L。等。使用监督机器学习技术开发的预测模型研究中的偏见风险:系统评价。BMJ 375,N2281(2021)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Mercer,D。K.等。抗菌肽的抗菌敏感性测试以更好地预测功效。正面。细胞。感染。微生物。 10,326(2020)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Wiegand,I.,Hilpert,K。&Hancock,R。E. W.琼脂和肉汤稀释方法,以确定抗菌物质的最小抑制浓度(MIC)。纳特。原始 3,163 - 175(2008)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Cesaro,A.,Torres,M。D. T.&de la Fuente-Nunez,C。肽抗生素设计和表征的方法。方法酶。 663,303 326(2022)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Powell,M。F.等。药物开发中的肽稳定性。ii。单氨基酸取代和糖基化对人血清中肽反应性的影响。Pharm。res。 10,1268年1273(1993)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Torres,M。D. T.等。可涂装和防抗性离子液体,用于消除病原体。ACS纳米 15,966 - 978(2021)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Pletzer,D.,Mansour,S。C.&Hancock,R。E. W.在鼠类埃斯卡普病原体引起的鼠皮下脓肿模型中,常规抗生素和抗生物胶质膜肽之间的协同作用。PLOS病原体。 14,E1007084(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Scheinpflug,K.,Krylova,O。&Strahl,H。通过Laurdan GP测量细胞膜流动性:荧光光谱和显微镜。方法摩尔。生物。 1520,159 - 174(2017)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Grage,S。L.,Afonin,S.,Kara,S.,Buth,G。&Ulrich,A。S.膜稀薄和由膜活性两亲性肽诱导的变薄和增厚。正面。细胞开发。生物。 4,65(2016)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Haney E.化学物理。脂质 163,82 - 93(2010)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Yan,J。等。两次命中率要比一个命中更好:NK-18的NK-18的膜活性和DNA结合相关的双重作用机制,这是一种源自哺乳动物NK-赖氨酸的新型抗菌肽。抗小动物。代理化学者。 57,220â228(2013)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Rokitskaya,T。I.,Kolodkin,N。I.,Kotova,E。A.&Antonenko,Y。N. Indarcidin对膜通透性的作用:载体机理与孔形成。生物基。生物。acta 1808年,91年(2011年)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Chan,D。I.,Prenner,E。J.&Vogel,H。J.色氨酸和富含精氨酸的抗菌肽:作用的结构和机制。生物基。生物。acta 1758年,1184年1202(2006)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Khandelia,H.,Ipsen,J。H.和Mouritsen,O。G.肽对脂质膜的影响。生物基。生物。acta 1778年,1528年1536年(2008年)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
帕特森(D.Proc。纳特。学院。科学。美国 114,E8324 e8332(2017)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Finger,S.,Kerth,A.,Dathe,M。&Blume,A。三价环状六肽在PG/PE膜中诱导脂质聚类的功效与其抗菌活性相关。生物基。生物。acta 1848年,2998年3006(2015)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Schmidt,N。W.&Wong,G。C. L.抗菌肽和诱导的膜曲率:几何形状,协调化学和分子工程。Curr。意见。固态母校。科学。 17,151 - 163(2013)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Zemel,A.,Ben-Shaul,A。&May,S。通过互动吸附的两亲性肽对脂质膜的自发曲率和弯曲刚度的调节。J. Phys。化学b 112,6988年6996(2008)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
Conibear,A。C.,Rosengren,K。J.,Daly,N。L.,Henriques,S。T.&Craik,D。J.α-defensin中的环状囊菌阶梯对于结构和稳定性很重要,但不是抗菌活性。J. Biol。化学 288,10830 10840(2013)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Torres,M。D. T.等。结构功能引导的抗微生物肽Polybia-CP识别活性决定因素并产生合成的治疗候选物。社区。生物。 1,221(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Lâzãr,V。等。抗生素耐药细菌表现出对抗菌肽的广泛核心敏感性。纳特。微生物。 3,718 - 731(2018)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Boaro,A。等。与WASP毒液衍生的抗感染活性的合成肽的结构功能引导的设计。细胞代表物理。科学。 4,101459(2023)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Lâzãr,V。,Snitser,O。,Barkan,D。&Kishony,R。抗生素组合减少金黄色葡萄球菌清除。自然 610,540 -546(2022)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Grâ©Zal,G。等。在抗生素耐药性的细菌演化后,转录组的可塑性和刻板印象重新布线。摩尔。生物。Evol。 40,MSAD020(2023)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
Sheard,D.E.,Oâbrien-Simpson,N。M.,Wade,J。D.&Sypovic,F。通过将抗生素与抗菌肽组合结合来对抗细菌耐药性。纯应用。化学 91,199 - 209(2019)。
文章一个 CAS一个 Google Scholar一个
Al Shaer,D.,Al Musaimi,O。,Albericio,F。&De la Torre,B。G. 2023 FDA潮汐(肽和寡核苷酸)收获。药品 17,243(2024)。
Silveira,G。G. O. S.等。抗生素肽:相关的临床前动物感染模型和翻译潜力。ACS Pharmacol。翻译。科学。 4,55 - 73(2021)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Silva,O。N.等。将肽毒素从黄蜂毒素重新利用为具有双重抗微生物和免疫调节特性的抗感染物。Proc。纳特。学院。科学。美国 117,26936 26945(2020)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Arquâ©,X。等。使用抗菌微动物和纳米运动员在体内对细菌感染的自主治疗。ACS纳米 16,7547年7558(2022)。
文章一个 PubMed一个 PubMed Central一个 Google Scholar一个
De Los Santos,L。等。聚生肽靶标克雷伯氏菌肺炎多糖塌陷生物膜。细胞代表物理。科学。 5,101869(2024)。
文章一个 Google Scholar一个
Stokes,J。M。等。抗生素发现的深度学习方法。细胞 180,688 - 702.E13(2020)。
文章一个 CAS一个 PubMed一个 PubMed Central一个 Google Scholar一个
Ribeiro,M。T.,Singh,S。和Guestrin,C。我为什么要信任您?:解释任何分类器的预测。在Proc。第22届ACM SIGKDD国际知识发现与数据挖掘会议1135年1144年(计算机协会,2016年)。
Lundberg,S.M。&Lee,S.-I。解释模型预测的统一方法。在Proc。第31届国际神经信息处理系统会议4768年4777(Curran Associates,2017年)。
Torres,M。D. T.等。用于抗生素优化的生成人工智能方法。预印本Biorxiv https://doi.org/10.1101/2024.11.27.625757(2024)。
Mirdita,M。等。群集和深层注释的蛋白质序列和比对的Uniclust数据库。核酸res。 45,D170 d176(2017)。
文章一个 CAS一个 PubMed一个 Google Scholar一个
