人工智能和生物分子预测软件时代的神经心理药理学
作者:Castro, Daniel C.
设计新型生物分子的经典方法
1865年,Crum-Brown和Fraser提出了结构活性关系(SARS)的观念[1]。SARS表征了化学结构与其预测的生物学活性之间的关系,从而为科学家提供了一个针对生物和化学分子的功效和效力的框架。通过实验改变这些元素,我们可以设计利用或避免特定治疗结果的分子(即降低细胞毒性或增加效力)。该理论的现代应用整合了化学分子与其生物学活性之间的数学关系,称为定量结构活性关系(QSARS)[2]。随着时间的流逝,QSAR演变成复杂的机器学习(ML)的技术,从而提取大量数据集,从而成为药物发现中的重要组成部分[3]。这些生物分子的定量进步继续向各个方向移动,并扩大了当今的生物技术景观。由于这些突破,我们现在能够将这些技术应用于研究蛋白质和其他生物分子,从而改变了我们处理神经心理药理学的方式。
氨基酸的串可以分为多种类别,每个类别都有助于定义分子的结构成分或生理作用。所得的蛋白质提供了一种复杂的构象,可以具有许多功能迭代,每种都可能与其他蛋白质或信号分子相互作用[4]。这些相互作用的确切性质在很大程度上取决于蛋白质折叠和展开,从而代表了活生物体中生物分子活性的关键机制[5]。但是,尽管蛋白质折叠本身被认为是细胞信号的必不可少的,但我们的模拟,预测和最终开发出新颖化合物以模仿这些过程的能力变得更加困难。从结构的角度来看,人工智能(AI)的使用使我们能够以高准确性的高度发展3D蛋白质结构[6,,,,7]。自从第一次尝试使用塑料模型可视化蛋白质分子以来,这项工作就发展起来。尽管塑料建模能够可视化折叠,但其效用仅限于具有简单结构的蛋白质。X射线晶体学和电子显微镜弹射了可视化到使用计算[8]。尽管对于理解孤立的蛋白质有用,但仍未解决蛋白质如何在与其他生物活性化合物的相互作用中变化。这就是为什么几十年来在上个世纪产生蛋白质模型的原因。鉴于他们可以假设的众多三级结构或形状,无法想象氨基酸如何结合和折叠[9]。
生物分子预测软件的出现
人工智能开发提高了机器学习(ML)和深度学习能力,提供了正在改变科学的强大算法。ML是AI的子场,通过在广泛的数据集中识别模式来工作,从而使系统可以学习和改进[10,,,,11]。尽管它要求程序员设置定义的任务,但它不需要明确的逐步指令。相反,ML模块允许系统学习和解释生物信息学数据,以改善过去的模型[12]。此过程允许该程序训练现有数据并大大减少错误。深度学习(DL)是ML的子层,采用神经网络(受动物大脑启发的结构和功能分析层)来处理非结构化数据(即不完整或多样化的信息),同时执行复杂的任务[13]。优化的DL调整了参数以滤除不必要的信息,可以实现可能或可能没有预定形状或功能的蛋白质结构设计。这使该程序可以考虑各种特征,例如在蛋白质构象中起着至关重要的作用的物理能力。
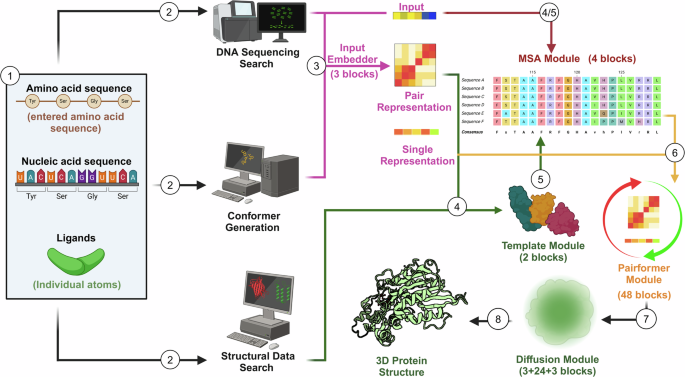
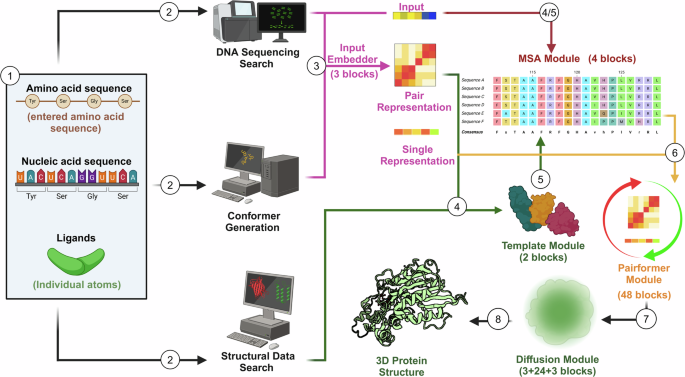
DL导致了一个名为Alphafold的人工智能程序的发展,该程序旨在解决蛋白质折叠并预测3D蛋白质结构[14,,,,15]。There are two previous versions of AlphaFold: AlphaFold 1 and AlphaFold 2. AlphaFold 1 was released in 2018, using neural networks to predict protein structures, while AlphaFold 2, released in 2021, switched its ML algorithm to continuously refine locations of nearby amino acids for every new input, and could predict structures with atomic level accuracy [16,,,,17]。尽管Alpha Fold 2可以对蛋白质结构进行建模及其相互作用,但它缺乏预测涉及更广泛生物分子的复合物的能力[18,,,,19,,,,20]。该软件的最新版本Alphafold 3(AF)在2024年发布,可以推断和模拟蛋白质 - 蛋白质相互作用,蛋白质 - 配体相互作用,小分子,核酸,离子和修饰的残基[21]。AF通过被喂养的分子列表来学习和估计置信范围,该值在该值中近似于实际结构。然后,它将用户输入的序列与数据库中的已知蛋白质结构(即蛋白质数据库)中的序列进行比较,以使分子数据与现有模板保持一致。一旦AF了解使用的生物分子框架,它就会激活其独特的扩散网络。Af的扩散网络会产生可能的构象,并通过应用化学和物理极限(例如分子力和键长)提供高度准确的分子结构[22]。它使用模板来指导新蛋白质的折叠过程,从而估计序列(然后是复合物)在组装后看起来的样子。AF不依赖同源性建模(相关蛋白的外观)来进一步可视化生物分子复合物,因为其编程识别模式,渗透生化特性并预测一级生物分子序列中的1D,2D和3D结构。它通过成功渲染图形表示而无需使用模板结构,同时预测先前未知的蛋白质折叠,从而完全绕过同源性[23]。
过去构建蛋白质预测工具不是为了分析物理和立体化学力相互作用的潜在情况[24]。当体现复合物可以完全改变构象甚至兼容时,承认物理和立体化学趋势至关重要[7]。AF通过拥有基于DL的结构预测体系结构来解决这一困难,该结构已接受了对生物分子结构的培训以预测其相互作用(图。 1)。AF以数学形式使用估算的生物分子来嵌入描述性信息,因为氨基酸和核酸(分别是蛋白质和DNA/RNA结构)之间存在不同的结构[25,,,,26]。AF还保持逼真的键几何形状,最大程度地减少空间冲突,并说明物理能量分布以实现原子结构的保真度[27]。这些原则告诉其预期物理学的限制可能会破坏形成分子结构的原子的空间排列(立体化学一致性)。此外,它可以防止不可能的配置在一定程度上从其学到和训练的同一立体化学原理中绘制。随着这些进展,AF的AF预测将如何进行复杂的折叠,因为它需要考虑的潜在操作[28,,,,29]。
Alphafold 3可以预测蛋白质,DNA/RNA分子和配体的结构。1)输入序列后,2)该软件同时使用多个数据库,以评估潜在的遗传序列,构象原型和结构构型。3)然后,输入嵌入器使用测序和顺符信息来编码并生成一个复合材料,从而导致单个和配对表示。4)然后,模板模块将从结构数据搜索获得的已知结构集成到对对表示中。4/5)在串联中,多个序列比对(MSA)模块将测序,配对表示和模板模型融合到迭代构建新型基本模板中。6)从这里开始,PairFormer模块使用MSA信息来测试不同的相互作用元素,完善预测的分子相互作用并反复更新配对和单一表示。7)最后,扩散模块将噪声应用于算法中,以改善局部立体化学和全局结构。
AF的另一个特征是它使用生物学驱动的原理来指导和识别关键模型特征。首先,AF筛选多个序列比对(MSA)识别物种之间的保守残基(氨基酸)[30]。此步骤有助于预测结构和功能重要的区域。当确定这些序列中进化的共同位置时,它们可以指出潜在的物理相互作用,以指导折叠和结合预测。共同发挥作用的复合物通常会一起发展,但这可以以多种方式发生[31]。识别融合进化点的复合物进化点使我们能够突出相关的突变。这些突变会影响生物分子如何与界面上的神经细胞和受体相互作用,在复杂结合中差异可能会带来意外后果。在测试生物分子如何达到构象状态时,对结构和功能区域的模块化适应有助于AF预测新的折叠模式,即使直接同源物未知。总而言之,AF继续以较早和类似的迭代为基础,但独特地融合了一种更灵活的设计,可以为药物发现提供令人兴奋的新机会。
生物分子预测软件的当前应用
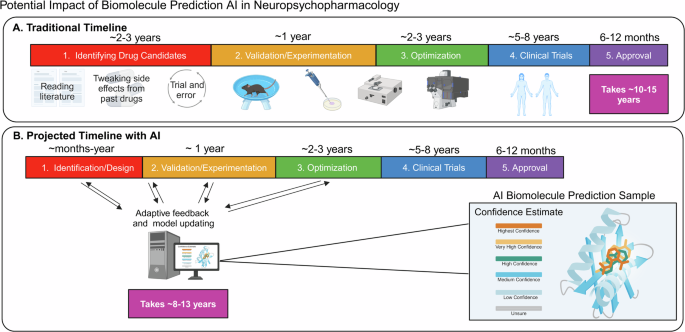
神经精神疾病研究通过具有改善靶标建模和药物开发的能力而受益于AF。更具体地说,AF可以通过分析候选药物来缩短时间窗口到毒性和不良事件的脱发生物分子(图。 2),协助新型G蛋白偶联受体(GPCR)的隔离和表征,以及在不久的将来授权时,可能会在生物分子复合物折叠期间(即错误折叠)期间预期意外问题[32,,,,33,,,,34]。目前,AF模型的表现优于传统建模(即同源性)工具的时间约70%,在硬靶标(没有已知同源物的蛋白质)上表现良好。在蛋白质结构预测(CASP)竞争的批判性评估中,已经观察到了其在该领域优势的证据。在这里,对候选预测软件进行了多个读数和类别的评估,例如全球距离测试(GDT)。GDT评估了预测的蛋白质结构与实验确定的结构的距离,从而提供了地面真相比较。随着时间的流逝,AF的得分提高了,最近2020年的竞赛得分与实验确定的结构相当[35]。
AF行动中AF的一个现实世界示例涉及发现痕量胺相关受体1(TAAR1)的配体,这是一种G蛋白耦合受体,它具有未知结构,是治疗神经精神疾病的有希望的靶标,从精神分裂症到药物使用障碍[32]。在中枢神经系统和外围发现TAAR1受体[36,,,,37]。TAAR1调节单胺能系统,尤其是多巴胺,5-羟色胺和去甲肾上腺素。与阻止D2多巴胺受体的常规抗精神病药不同,TAAR1激活可以间接调节多巴胺能的张力,从而减少了多巴胺能(中央多巴胺能传播的持续增加)而不会引起运动副作用,例如迟发性运动障碍(Tardive Dyskinesia)(常见的抗精神病学副作用)。在外围,新兴的证据表明TAAR1激活与胰岛素分泌有关(胰腺)[38],能量稳态和体重调节,同时调节神经回路[39]。这引起了对TAAR1的兴趣,作为2型糖尿病和肥胖症等代谢疾病的潜在靶标。考虑到其广泛的效用,人们对开发化合物调节该受体有浓厚的兴趣。不幸的是,缺乏高分辨率的结构数据阻碍了像TAAR1这样的GPCR的药物发现。尽管这通常是传统,基于同源的方法的主要障碍,但AF能够产生TAAR1的高信心结构模型,从而使研究人员能够加快基于结构的虚拟筛查。研究人员在计算上停靠了超过1600万个片段样的分子,从锌数据库中置于TAAR1的估计结合位点。AF衍生的结构确定了680万种能够在结合位点进行对接的化合物,从中选择了30个顶级候选者进行实验测试。AF筛网中最有效,最有选择性的化合物,称为化合物65,对其他AMINEGIC受体和有利的药代动力学特性表现出良好的选择性。在鼠标模型中使用体内进一步验证了化合物65。他们发现,化合物65调节的体温并产生了感觉运动门控行为(全身退缩),这是抗精神病药样作用的动物模型。这些作用在TAAR1敲除小鼠中不存在,证实观察到的药理学是通过TAAR1激活介导的。在此过程中,对基于同源的模型进行了基准药物发现功效的模型进行了测试。他们发现,与同源性模型的22%的命中率相比,AF衍生的模型的命中率达到60%,高三倍。诸如此类的案例突出了新一代预测软件的优势,尤其是与药物发现的早期阶段有关。
生物分子预测软件的当前局限性
显然,生物分子预测软件代表了生物医学AI技术的重要飞跃[40]。准确预测结构并在统一框架中检查其相互作用将减少时间范围以搜索或设计新药候选者。但是,这些模型仍然有几个关键的领域,在这些领域中,持续发展是必要的,例如它们创建虚假的结构命令的优势(也称为幻觉[[41])。这些数字幻觉是预测的结构特征,可能在模型中看起来很好和自信,但实际上并不存在于蛋白质的真实本机结构中,通常是由于模型培训或输入数据中的局限性或工件所致。解决这些问题和类似问题(例如,不完整/混乱的生物信息学数据)将有助于减轻未来模型中的这些破坏,因为人类干预会在有助于准确的模拟中为指导提供指导。
AF擅长预测结构之间的相互作用(即配体和受体之间),但不能量化亲和力或效能(即该相互作用的效果)。为了评估这一点,需要其他建模工具或实验数据。因此,最好将AF视为用于进一步计算或湿LAB工作的高分辨率输入,而不是预测药物功效的独立解决方案。相关的是,AF还努力融合可能影响分子相互作用的细胞外空间条件。当算法试图预测分子相互作用时,处理手性和手性分子(原子的结合和排列)时,准确性会下降。21,,,,23]。它整合了一些基本的化学,但是到目前为止,还没有推断必需的空间分布来组装可以在实验室中很容易或现实合成的分子[14,,,,42,,,,43]。结果,它通常会省略像血红素基团,虚假陈述金属配位位点这样的关键组成部分,并且无法说明共价药物结合或酶促活性位点的几何形状。
最后,在对内部无序区域,非特异性蛋白质相互作用以及具有显着构象柔韧性的蛋白质进行建模时,AF会表现出局限性。尽管试图从图像产生(例如扩散模型)中融合AI技术,但AF仍然可以产生结构上令人难以置信的特征,例如原子重叠或空间冲突[44]。它还偶尔会省略关键的结构要素,因此需要专家人类校正。对于有效的药物设计,AF将受益于从药物化学,结构生物学和物理学中更好地整合原理,从而更准确地反映现实世界的分子力和对接行为。人类的监督对于指导这些模型并在现实的生化环境中解释结果至关重要。最终,必须通过实验测定来验证预测,并建议研究人员生成多个模型并比较输出以提高可靠性并确定一致的结构特征。
讨论
诊断,生物分子预测软件可以提供有关遗传突变如何影响蛋白质结构和相互作用的见解[45]。通过了解离散突变如何影响蛋白质功能,我们可以更好地设计解释这些功能变化的分子。尽管如此,尽管AI技术可以提供有用的建议,但分子的体内测试仍然是指导选择候选临床试验药物的金标准。在测试模型生物如何应对旨在针对特定部位的治疗作用的生物分子的反应[46]。但是,在预测软件的指导下,后端药物开发可以显着改善和加快。展望未来,有必要消除软件开发人员和软件用户之间的现有断开连接。现实的护栏在AI的开发和部署中至关重要,必须采取关键的预防措施来保护技术可以干预和执行人类当前处理的功能的程度。仅当神经心理药理学研究中的数字素养现代化以实施这些类型的工具时,才能实现这一点。作为一个跨学科的领域,神经心理药理学将从优化的新型药物发现,复杂的生物行为建模以及标准化但基于精确的治疗方法中显着受益。在服用和接受治疗的人中,AI对神经精神病治疗的采用将是一个困难但很重要的对话。但是,我们认为,关于如何以及何时纳入创新的AI技术的公开讨论代表了精确医学方面的激动人心的一步。
引文多样性声明
作者证明,他们努力在选择本文中使用的引用时要注意多样性。
参考
棕色AC,Fraser Tr。关于化学构成与生理作用之间的联系;特别是指源自史氏菌,布鲁西亚,贝巴里亚,codeia,morphia和Nicotia的铵碱的生理作用。J Anat Physiol。1868; 2:224 42。
CAS一个 PubMed Central一个 Google Scholar一个
Hansch C,Fujita T. p-ï-分析。生物活性和化学结构相关的一种方法。J Am Chem Soc。1964; 86:1616 26。
文章一个 CAS一个 Google Scholar一个
Soares ta,Nunes-Alves A,Mazzolari A,Ruggiu F,Wei GW,Merz K.定量结构活动关系(QSAR)研究的(重新)由机器学习方法的激增推动。J Chem Inf模型。2022; 62:5317 20。
文章一个 CAS一个 Google Scholar一个
Alberts B,Johnson A,Lewis J,Raff M,Roberts K,Walter P等。蛋白质的形状和结构。在细胞的分子生物学中。第四版。花环科学;纽约;2002https://www.ncbi.nlm.nih.gov/books/nbk21054/。Dãaaz-Villanueva JF,Dãaaz-Molina R,Garcãa-GonzãlezV。蛋白质折叠和蛋白质的机制。
Int J Mol Sci。2015; 16:17193 230。
文章一个 PubMed Central一个 Google Scholar一个
Vilar S,CostanziS。通过QSAR分析和基于对接的评分来预测生物学活动。方法摩尔生物。2012; 914:271 84。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Blanco-GonzãlezA,Cabezãna,Seco-GonzãlezA,Conde-Torres D,Antelo-Riveiro P,Pié±Eiroâ,Garcia-FandinoR。AI在药物发现中的作用:挑战,机遇和策略。药品。2023; 16:891。
文章一个 PubMed Central一个 Google Scholar一个
Boumedine N,BouroubiS。使用杜鹃搜索与爬山算法的组合搜索中的3D晶格HP模型中的蛋白质折叠。应用软计算。2022; 119:108564。
文章一个 Google Scholar一个
Kuhlman B,Bradley P.蛋白质结构预测和设计的进步。Nat Rev Mol Cell Biol。2019; 20:681 97。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Mosavi A,Salimi M,Ardabili SF,Amidpour M,Rabczuk T,Shamshirband S等。能源系统中机器学习模型的艺术状态,系统评价。能量。2019; 12:1301。
文章一个 Google Scholar一个
Schauperl M,Denny RA。基于AI的药物发现中的蛋白质结构预测:影响和挑战。J Chem Inf模型。2022; 62:3142 - 56。
文章一个 CAS一个 Google Scholar一个
Franã§ARP,Borges Monteiro AC,Arthur R&Iano Y第3章 - 现代数字时代大数据,图像和信号处理中深度学习的概述。在Piuri V,Raj S,Genovese A&Srivastava R,编辑中。深度学习方法的趋势。学术出版社;2021。63â87。https://doi.org/10.1016/b978-0-12-82226-3.00003-9。Abdolrasol MGM,Hussain SMS,Ustun TS,Sarker MR,Hannan MA,Mohamed R等。
基于人工神经网络的优化技术:评论。电子产品。2021; 10:2689。
文章一个 Google Scholar一个
Jumper J,Evans R,Pritzel A,Green T,Figurnov M,Ronneberger O,Tunyasuvunakool K.具有Alphafold的高度精确蛋白质结构预测。自然。2021; 596:583 9。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Thornton JM,Laskowski RA,Borkakoti N. Alphafold预示着生物学和医学的数据驱动革命。Nat Med。2021; 27:1666 9。
文章一个 CAS一个 Google Scholar一个
Alquraishi M.蛋白质结构预测发生了革新。自然。2021; 596:487 - 8。
文章一个 Google Scholar一个
Callaway E. DeepMind的AI预测了大量蛋白质的结构。自然。2021; 595:635。
文章一个 CAS一个 Google Scholar一个
Skolnick J,Gao M,Zhou H,Singh S. Alphafold 2:为什么它起作用及其对理解蛋白质序列,结构和功能的关系的影响。J Chem Inf模型。2021; 61:4827 31。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Nussinov R,Zhang M,Liu Y,Jang H. Alphafold,人工智能(AI)和变构。J Phys Chem B. 2022; 126:6372 - 83。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Ma W,Zhang S,Li Z,Jiang M,Wang S,Lu W,Bi X,Jiang H,Zhang H,Wei Z。通过利用Alphafold预测的蛋白质结构来增强蛋白质功能预测性能。J Chem Inf模型。2022; 62:4008 17。
文章一个 CAS一个 Google Scholar一个
Abramson J,Adler J,Dunger J,Evans R,Green T,Pritzel A,Ronneberger O.与Alphafold 3。自然相互作用的准确结构预测。2024; 630:493 500。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Roy R,Al-Hashimi HM。AlphaFold3朝着解码分子行为和生物学计算迈出了一步。NAT结构摩尔生物。2024; 31:997 1000。
文章一个 CAS一个 Google Scholar一个
Desai D,Kantliwala SV,Vybhavi J,Ravi R,Patel H,Patel J. Alphafold的评论3:药物设计和治疗剂的变革性进步。肉质。2024; 16:e63646。
Bertoline LMF,Lima AN,Krieger JE,Teixeira SK。Alphafold2之前和之后:蛋白质结构预测的概述。前生物信息。2023; 3:1120370。
文章一个 PubMed Central一个 Google Scholar一个
Anusha,Zhang Z,Li J,Zuo H,Mao C. Alphafold 3辅助DNA图案的设计,以组装三角形。J Am Chem Soc。2024; 146:25422 - 5。
文章一个 CAS一个 Google Scholar一个
Wee J,Wei G-W。评估Alphafold 3的蛋白蛋白复合物,以预测突变时结合自由能的变化。J Chem Inf模型。2024; 64:6676 83。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Yang Z,Zeng X,Zhao Y,Chen R. Alphafold2及其在生物学和医学领域的应用。信号转移目标。2023; 8:1 14。
与AlphaFold2相比,Hoffmann F Alphafold3及其改进。中等的。2024。https://medium.com/@falk_hoffmann/alphafold3-and-ismprovements-in-comparison-to-alphafold2-96815ffbb044。Thompson B,PetriäHoweN。Alphafold3.0:AI蛋白预测器进行升级。
自然。2024。https://doi.org/10.1038/d41586-024-01385-x
文章一个 PubMed Central一个 Google Scholar一个
沃斯尼格(Wossnig),l alphafold3 - 计算药物发现的下一步是什么?中等的。2024。https://medium.com/@leowosnig/alphafold3-whats-next-in-computations-drug-discovery-2da534c0845e。Lu X,Kensche PR,Huynen MA,Notebaart RA。
基因组进化预测了蛋白质复合物中的遗传相互作用,并揭示了癌症药物靶标。纳特社区。2013; 4:2124。
文章一个 Google Scholar一个
Dãaaz-HolguãnA,Saarinen M,Vo DD,Sturchio A,Branzell N,Cabeza de vaca I,Hu H. Alphafold加速了针对痕量胺相关受体的精神激动剂的发现。2024; 10:EADN1524。
文章一个 PubMed Central一个 Google Scholar一个
X-H,Li J-R,Shen S-Y,Xu He。AlphaFold3与实验结构:评估配体结合G蛋白偶联受体的精度。Acta Pharmacol Sin。2024; 46:1111 22。https://doi.org/10.1038/s41401-024-01429-y
文章一个 CAS一个 Google Scholar一个
Krokidis MG,Koumadorakis DE,Lazaros K,Ivantsik O,Exarchos TP,Vrahatis AG,Kotsiantis S. Alphafold3:应用程序和绩效见解的概述。Int J Mol Sci。2025; 26:3671。
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Callaway E. ‘It will change everything’: DeepMind’s AI makes gigantic leap in solving protein structures.自然。2020;588:203–4.
文章一个 CAS一个 Google Scholar一个
Nair PC, Chalker JM, McKinnon RA, Langmead CJ, Gregory KJ, Bastiampillai T. Trace amine-associated receptor 1 (TAAR1): molecular and clinical insights for the treatment of schizophrenia and related comorbidities.ACS Pharmacol Transl Sci.2022;5:183–8.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Dedic N, Dworak H, Zeni C, Rutigliano G, Howes OD.Therapeutic potential of TAAR1 agonists in schizophrenia: evidence from preclinical models and clinical studies.Int J Mol Sci。2021;22:13185.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Michael ES, Covic L, Kuliopulos A. Trace amine-associated receptor 1 (TAAR1) promotes anti-diabetic signaling in insulin-secreting cells.J Biol Chem。2019;294:4401–11.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Dedic N, Wang L, Hajos-Korcsok E, Hecksher-Sørensen J, Roostalu U, Vickers SP, et al.TAAR1 agonists improve glycemic control, reduce body weight and modulate neurocircuits governing energy balance and feeding.mol代谢。2024;80:101883.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Arnold C. AlphaFold touted as next big thing for drug discovery — but is it?自然。2023;622:15–17.
文章一个 CAS一个 Google Scholar一个
Lowe, D AlphaFold 3 debuts.2024。https://www.science.org/content/blog-post/alphafold-3-debuts。Tunyasuvunakool K, Adler J, Wu Z, Green T, Zielinski M, ŽÃdek A, Bridgland A, Cowie A, Meyer C. Highly accurate protein structure prediction for the human proteome.
自然。2021;596:590–6.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Steinkellner G, Kroutil W, Gruber K, Gruber CC.AlphaFold 3 is great — but it still needs human help to get chemistry right.自然。2025;637:548.
文章一个 CAS一个 Google Scholar一个
Brotzakis ZF, Zhang S, Murtada MH, Vendruscolo M. AlphaFold prediction of structural ensembles of disordered proteins.纳特社区。2025;16:1632.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
Yao M, Miller GW, Vardarajan BN, Baccarelli AA, Guo Z, Liu Z. Deciphering proteins in Alzheimer’s disease: a new mendelian randomization method integrated with AlphaFold3 for 3D structure prediction.Cell Genom.2024;4:100700.
文章一个 CAS一个 PubMed Central一个 Google Scholar一个
van der Lee M, Swen JJ.Artificial intelligence in pharmacology research and practice.Clin Transl Sci。2022;16:31–36.
致谢
All figures were created with Biorender.com.
资金
All this work has been supported by funding to R.A.G.R.from the Howard Hughes Medical Institute (HHMI) Gilliam Fellowship (GT17052) and the Washington University National Institute of Health Initiative for Maximizing Student Development Program Grant (5T32GM148405);L.N.M.Q.is funded by the Washington University Neuroscience Postbaccalaureate Research Education Program (NeuroPREP) Program (R25NS130965);H.R. is funded by the Washington University Vagelos Undergraduate Research Fellowship (Roy and Diana Vagelos Division of Biology and Biomedical Sciences);D.C.C.is funded by the National Institute of Mental Health (5R01MH132504), the National Institute on Drug Abuse (5R00DA049862), the McDonnell Center for Cellular Molecular Neurobiology, the McDonnell Center for Systems Neuroscience, and the Diabetes Research Center (P30 DK020579)at Washington University School of Medicine in St. Louis.作者信息
作者宣布没有竞争利益。
附加信息
Publisher’s note
关于已发表的地图和机构隶属关系中的管辖权主张,Springer自然仍然是中立的。
Rights and permissions开放访问
This article is licensed under a Creative Commons Attribution 4.0 International License, which permits use, sharing, adaptation, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons licence, and indicate if changes were made.
The images or other third party material in this article are included in the article’s Creative Commons licence, unless indicated otherwise in a credit line to the material.If material is not included in the article’s Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder.要查看此许可证的副本,请访问http://creativecommons.org/licenses/4.0/。重印和权限
引用本文
GarcÃa-Reyes, R.A., Massó Quiñones, L.N., Ruy, H.
等。Neuropsychopharmacology in the era of artificial intelligence and biomolecule prediction software.NPP—Digit Psychiatry Neurosci3 , 16 (2025).https://doi.org/10.1038/s44277-025-00038-9
已收到:
修改:
公认:
出版:
doi:https://doi.org/10.1038/s44277-025-00038-9